AllStars Negative Control siRNA
RNAi实验的阴性对照
RNAi实验的阴性对照

Cat. No. / ID: 1027281

Cat. No. / ID: 1027280
AllStars Negative Control siRNA是目前经过最全面的测试和验证的阴性对照siRNA。该siRNA与已知的哺乳动物基因无同源性。已经运用Affymetrix GeneChip芯片和多种细胞实验验证了其有效性,确保该siRNA对基因表达和表型的非特异性效应最小。通过比较基因特异性siRNA和阴性对照,可以得出靶基因敲减对基因表达和表型的影响,而最小的非特异性效应确保这一结论真实可靠。如果阴性对照引起非特异性效应,则RNAi实验结果会被干扰且难以解释。克隆实验证实AllStars Negative Control siRNA进入RISC。AllStars Negative Control siRNA正处于专利申请中,该序列是专有的。
表中实验用来确证AllStars Negative Control siRNA的性能特征。
| 实验类型 | 实验名称 | 目的 | 使用AllStars Negative Control siRNA的结果 |
|---|---|---|---|
| 全基因分析 | Affymetrix GeneChip芯片 | 非特异性调控基因表达 | 最少数目的基因被调控 |
| 细胞水平分析 | 活细胞核染色 | 核大小 | 正常 |
| 细胞水平分析 | 细胞数目 | 增殖速率 | 无变化 |
| 细胞水平分析 | 核酸整合 | DNA合成率 | 无变化 |
| 细胞水平分析 | 活细胞染色排除 | 细胞毒性作用 | 无变化 |
| 细胞水平分析 | DNA染色 | 细胞周期分布 | 正常 |
| RISC整合分析(HeLa和MCF-7细胞) | 报告质粒转染 | 确定siRNA是否整合入RISC(一个有效的阴性对照应整合入RISC) | 整合入RISC |
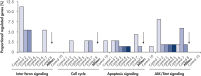
转染多种阴性对照siRNA后,运用全基因组分析检测它们对基因表达的非特异性效应。使用HiPerFect Transfection Reagent将不同来源的多种阴性对照siRNA转染至MCF-7、K562和原代HUVEC细胞。这些阴性对照siRNA包括非沉默siRNAs(与哺乳动物基因无同源性)、干扰siRNAs(碱基组成与基因特异性siRNA相同而碱基序列不同的siRNAs)和靶向人工合成报告基因的siRNAs。随后,用Affymetrix GeneChip芯片测试人全基因谱的表达。使用AllStars Negative Control siRNA,非特异性调控基因最少,适合做阴性对照。而其它的阴性对照siRNAs则导致许多源于细胞通路的基因的非特异性调控(参见" Low nonspecific effects on expression")。
活细胞核染色用于测量细胞核的大小。细胞核大小的变化可作为细胞周期紊乱或生长抑制的信号。我们测试了一系列不同类型的阴性对照siRNAs,AllStars Negative Control siRNA结果最好。与未转染的细胞相比,转染AllStars Negative Control siRNA不会改变细胞核的大小。相反,转染其他的阴性对照siRNAs会使细胞核增大(参见" Nuclear size phenotype unaffected")。
转染一系列阴性对照siRNAs后,进行细胞计数以确定细胞增殖是否正常。与未转染细胞相比,转染AllStars Negative Control siRNA后细胞数目几乎无差异。相反,转染其他的siRNAs后细胞数目显著下降,如Control 1,表明这些siRNAs会导致生长缺陷表型(参见" Cell number unaffected")。
通过检测未转染和转染了一系列不同阴性对照siRNAs的HCT-116细胞的核酸整合,以确定DNA合成率。通过检测溴脱氧尿嘧啶核苷(BrdU)的摄取来测量核酸整合,BrdU是一种在DNA复制过程中代替胸腺嘧啶并可整合进新合成的DNA的胸腺嘧啶碱基类似物。DNA合成率的改变预示着细胞生长和细胞周期的变化。使用AllStars Negative Control siRNA转染的细胞,BrdU整合率与未转染细胞非常相似。然而,使用其他阴性对照siRNAs转染细胞,DNA合成率则较低,这表明该siRNA影响了细胞生长和细胞周期(参见" Normal DNA synthesis phenotype")。
使用活细胞染色排除实验测一系列阴性对照siRNAs潜在的细胞毒性。结果显示,使用AllStars Negative Control siRNA转染的细胞与未转染细胞具有相同的活细胞数目。相反,其他的阴性对照siRNAs则导致细胞毒性增加(参见" No increase in cytotoxic effects")。
细胞固定后用DNA染色法检测细胞周期的分布(处于G1/G0期、S期和G2期的细胞数量)。细胞转染AllStars Negative Control siRNA后,处于各细胞周期的细胞数的比例与未转染的细胞相似(参见" Normal cell-cycle distribution")。这一结果表明AllStars Negative Control siRNA没有对细胞周期产生不利影响。
为了阴性对照RNAi实验的准确性,阴性对照siRNA应被整合到RISC(RNA诱导的沉默复合物)里。这意味着,阴性对照siRNA与基因特异性siRNA经过了相同的生物学过程,这使得我们能够比较使用基因特异性siRNA和阴性对照siRNA得出的两组数据,确信这是基因敲减的结果。
实验如下:
报告质粒和非互补siRNA共转染会导致荧光蛋白和His标签的强烈表达。当报告质粒和AllStars Negative Control siRNA共转染时,由它的互补序列将AllStars Negative Control siRNA的表达敲减,这使得整个编码荧光报告基因、His标签和siRNA目标序列的转录mRNA被降解。而mRNA的降解使得融合蛋白被敲减(参见" AllStars Negative Control siRNA is incorporated into RISC"和" Western analysis shows AllStars Negative Control siRNA enters RISC")。要实现对基因敲减的观测,必须将AllStars Negative Control siRNA整合到RISC。
 Nuclear size phenotype unaffected.
Nuclear size phenotype unaffected.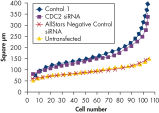 Cell number unaffected.
Cell number unaffected.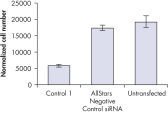 Normal DNA synthesis phenotype.
Normal DNA synthesis phenotype. No increase in cytotoxic effects.
No increase in cytotoxic effects. Normal cell-cycle distribution.
Normal cell-cycle distribution. Reporter construct for RISC-incorporation experiment
Reporter construct for RISC-incorporation experiment Western analysis shows AllStars Negative Control siRNA enters RISC.
Western analysis shows AllStars Negative Control siRNA enters RISC.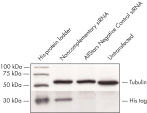 AllStars Negative Control siRNA is incorporated into RISC.
AllStars Negative Control siRNA is incorporated into RISC.
转染阴性对照siRNA是每个RNA干扰试验必不可少的。阴性对照结果应该与从未转染细胞得到的结果相比较。转染阴性对照siRNA和未转染细胞的基因表达和表现型应该完全相似。如果在转染阴性对照siRNA的细胞里发现了基因表达和表现型的改变,这种改变是由转染过程或siRNA毒性和序列的非互补性引起的非特异性改变。为了确保RNAi/miRNA结果的可靠性,非特异性效应应该最小。
阴性对照结果也能与正在研究的基因特异性的RNAi/miRNA结果进行比较。这种比较使得研究者能准确描述靶基因敲减对基因表达和表现型的影响,因为阴性对照siRNA只是RNAi/miRNA的序列不同,而经历的生物学过程是完全相同的。
AllStars Negative Control siRNA的结果可用于以下应用:
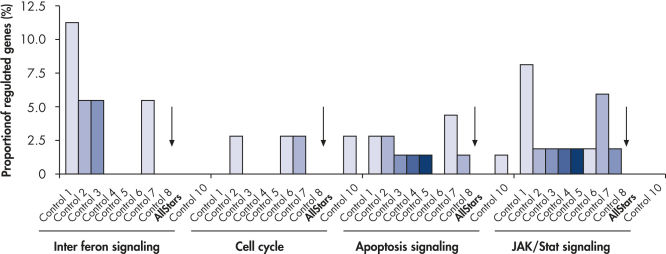
Multiple negative control siRNAs (Control 1– Control 10) were transfected in triplicate into MCF-7 cells. After incubation, cRNA was prepared and hybridized to Affymetrix human U133 GeneChip arrays. Regulated genes were identified as genes that showed at least a 1.5-fold change in expression (both upregulated and downregulated) compared to untransfected cells. Ingenuity pathway analysis software was used to determine the proportion of regulated genes in each pathway compared to the total number of genes identified as central to that pathway. Where a bar appears in the figure, this means that genes in the pathway were regulated by the siRNA. If every pathway gene was regulated, the relative proportion would be 100%. Lower bars therefore indicate a lower relative proportion of regulated genes within that pathway. Where no bar appears, no genes of the pathway were regulated by the siRNA. AllStars Negative Control siRNA (indicated with arrow) resulted in the lowest number of regulated genes. In contrast, other control siRNAs resulted in higher numbers of regulated genes from important cellular pathways.
| Features | Specifications |
|---|---|
| Design | Predesigned/validated by Affymetrix GeneChip Array and cell-based assays |
| Species | Human, mouse, rat |
| Scale or yield | 5 nmol, 20 nmol |
| Format | Tube |
| Target sequence provided | No |
| Modification | Yes |