EpiTect Hi-C Kit
염색질 폴딩(folding)의 고해상도 매핑, 유전체 서열의 고품질 어셈블리, 일배체형 페이징(phasing), 염색체 재배열 확인에 사용
염색질 폴딩(folding)의 고해상도 매핑, 유전체 서열의 고품질 어셈블리, 일배체형 페이징(phasing), 염색체 재배열 확인에 사용
✓ 연중무휴 하루 24시간 자동 온라인 주문 처리
✓ 풍부한 지식과 전문성을 갖춘 제품 및 기술 지원
✓ 신속하고 안정적인 (재)주문
Cat. No. / ID: 59971
✓ 연중무휴 하루 24시간 자동 온라인 주문 처리
✓ 풍부한 지식과 전문성을 갖춘 제품 및 기술 지원
✓ 신속하고 안정적인 (재)주문
Hi-C는 원래 유전체 전체 염색체 구조를 포착하는 강력한 기술로 고안되었으며, 염색질 폴딩을 kb 해상도로 특성 분석할 수 있게 해줍니다. 하지만 이 기술은 다른 중요한 응용 분야에서도 활용되고 있습니다. 예를 들어, Hi-C는 알려진 참조 유전체가 없는 유기체로부터 매우 길고 적은 수의 스캐폴드로 아주 연속적인 유전체 어셈블리를 생성하는 데 사용됩니다. 또한 Hi-C는 일배체형 페이징과 염색체 재배열 검출에도 매우 유용합니다.
EpiTect Hi-C Kit는 안정적이면서도 간단하고 빠른 프로토콜을 제공하며, 적은 세포 투입량 요건으로 2일 이내에 교차 결합된 세포로부터 고품질 Hi-C Illumina NGS 라이브러리를 생성할 수 있습니다.
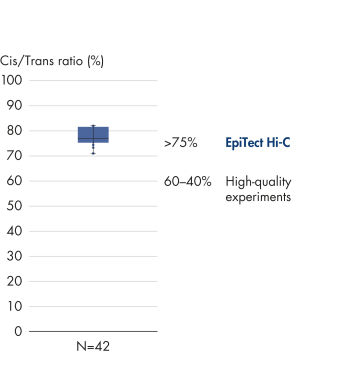
EpiTect Hi-C Kit는 고가의 다운스트림 심층 염기서열 분석에서 최고 수준의 데이터를 생성할 수 있도록 고품질 Hi-C NGS 라이브러리를 생성합니다. 키트의 성능을 평가하기 위해 40개 이상의 EpiTect Hi-C 라이브러리에서 얻은 염기서열 분석 결과를 분석했습니다. 가장 중요한 QC 지표는 다음 그림에 나와 있습니다. Hi-C 이벤트의 백분율, 장거리 cis 상호 작용의 백분율, Cis/Trans 비율, EpiTect Hi-C Kit 사용 시 가닥 방향 편향 없음, 단일 제한 단편에서 파생된 판독 쌍의 백분율. 데이터에 따르면 EpiTect Hi-C Kit는 평균적으로 성공적인 Hi-C 실험에 적합한 것으로 간주되는 기준을 훨씬 뛰어넘는 NGS 라이브러리를 생성합니다.
Hi-C는 유전체 전체 범위에서 염색질 상호 작용을 포착하는 근접 결찰(ligation) 분석입니다. EpiTect Hi-C Kit는 Illumina와 호환되는 NGS 라이브러리를 생성하는 특화된 DNA 준비 과정입니다(그림 EpiTect Hi-C 워크플로우 – 1일 차 및 EpiTect Hi-C 워크플로우 – 2일 차 참고). 간단히 설명하자면, 이 분석은 DNA 결합 단백질과 DNA의 화학적 가교 결합을 통해 염색질 구조가 동결된 핵을 정제하는 것으로 시작됩니다. 그런 다음 4bp 제한 효소로 DNA를 완전히 분해합니다. 열린 DNA 말단은 비오틴으로 라벨링되고 이후 라이게이션 됩니다. Hi-C 결찰(ligation) 생성물의 쌍단 염기서열 분석을 통해 공간적으로 밀접하게 연관된 DNA 가닥에서 파생된 매우 많은 수의 키메라 염기서열을 식별할 수 있습니다. 두 염기서열이 서로 연결될 가능성은 공간에서의 평균 거리의 함수로 계산됩니다. 결찰(ligation) 접합부의 정량화를 통해 염색질 폴딩의 고해상도 매핑을 달성할 수 있는 DNA 접촉 빈도를 측정할 수 있습니다.
EpiTect Hi-C 워크플로우(그림 EpiTect Hi-C 워크플로우 – 1일 차 및 EpiTect Hi-C 워크플로우 – 2일 차 참고)는 기존에 발표된 프로토콜에 비해 크게 개선되었습니다. 일주일이 걸리던 복잡한 절차가 단 1.5일이 소요되는 간단하고 안정적인 프로토콜로 바뀌었습니다. 또한 샘플 투입량 요건이 1/10로 줄어들어 단 5000개의 세포로 Hi-C NGS 라이브러리를 생성할 수 있습니다. 이 프로토콜은 포유류 세포 배양에서 얻은 가교 세포 작업을 위해 개발되었습니다.
EpiTect Hi-C 절차는 핵을 조심스럽게 정제하고 투과 처리(permeabilize)하여 초기 분해 및 결찰(ligation) 단계에서 유전체의 공간 구성을 유지하는 in situ(즉, 핵 내) Hi-C 방법의 한 버전입니다. 이 과정은 유전체 구성을 반영하지 않는 무정보 결찰(ligation) 이벤트로 인한 배경 노이즈를 최소화하기 위해 필수적입니다. 이는 온전한 핵이 가교 결합된 복합체의 이동과 무작위 충돌을 제한하여 그러한 결찰(ligation) 이벤트가 주로 위상학적으로 연결된 DNA 단편 사이에서 발생하기 때문입니다.
Hi-C NGS 라이브러리 구축하기
EpiTect Hi-C Kit 워크플로우는 2개의 부분으로 구성되며 각각 하루 안에 완료할 수 있습니다. 프로토콜의 단계는 아래 표에 요약되어 있으며 그림 EpiTect Hi-C 워크플로우 – 1일 차 및 EpiTect Hi-C 워크플로우 – 2일 차로 시각화되어 있습니다. 포함된 Illumina 어댑터에는 최대 6개의 샘플을 멀티플렉스 염기서열 분석할 수 있는 염기서열 바코드가 있습니다.
전체 프로토콜을 보려면 상세한 EpiTect Hi-C 안내서를 참고하십시오.
데이터 분석
Hi-C 데이터 분석은 GeneGlobe 데이터 분석 센터에서 제공됩니다. Hi-C 염기서열 분석 결과는 오픈 소스 도구를 사용하여 QC 염기서열 분석 보고서, Hi-C 접촉 매트릭스 및 염색질 접촉 맵의 시각화를 제공하는 EpiTect Hi-C 데이터 분석 포털을 사용하여 분석할 수 있습니다. 자세한 내용은 EpiTect Hi-C 데이터 분석 포털 사용자 가이드를 참고하십시오.
염색질 구조
Hi-C는 핵 구성 분석을 위한 매우 중요한 도구로 빠르게 자리 잡았습니다. Hi-C 데이터를 분석한 결과, 유전체를 염색체 영역, 염색체 하위 구획, 위상 결합 영역(Topologically Associated Domain, TAD), DNA 루프로 분할하는 여러 층의 공간 구성으로 이루어진 유전체 구조의 놀라운 복잡성이 밝혀졌습니다(그림 염색질 구성 수준 참고). 또한 유전체 구성은 역동적이며 발달 과정에서 변화합니다. 어떤 세포 유형에서도 염색체가 똑같이 폴딩되는 경우는 없습니다.
염색체 재배열 및 복제수 변이
개별 염색체는 물리적으로 별개의 영역에 분리되어 있으므로 Hi-C가 포착하는 DNA 상호 작용은 주로 같은 염색체(cis 내)의 DNA 사이에서 이루어지며 다른 염색체(trans 내) 간에는 상호 작용이 거의 이루어지지 않습니다. 이러한 현상 덕분에 Hi-C는 전좌 및 기타 관심 있는 구조적 변이를 파악하기 위한 유전체 전체 분석으로 사용할 수 있습니다. 다른 NGS 기술에 비해 Hi-C는 필요한 커버리지가 매우 적기 때문에 비용을 절감할 수 있습니다. 또한, 매핑이 잘 되지 않는 영역이 포함된 재배열은 표준 NGS 방법보다 Hi-C를 사용하여 더 잘 발견할 수 있습니다. 편리하게도, 동일한 Hi-C 데이터를 사용하여 복제수 변경을 확인할 수도 있습니다.
유전체 어셈블리 – 일배체형 페이징(phasing)
새로운 종의 유전체를 염기서열 분석하고 어셈블리할 때, 염기서열 스캐폴드 생성은 염기서열 분석 범위를 넘어서는 반복적인 염기서열로 인해 종종 제한을 받습니다. Hi-C 데이터에서 대부분의 상호 작용은 같은 염색체에 있는 유전자좌 사이의 cis 내에서 발생합니다. 또한 이러한 cis 상호 작용의 상당 부분은 수백만 개의 DNA 염기로 분리된 유전자좌 사이에서 발생하는 장거리 상호작용입니다. 이러한 염색질 상호 작용의 특성을 활용하여 참조 유전체(reference genome) 없이도 염기서열 스캐폴드를 거의 전체 길이에 가까운 염색체로 정렬, 배향, 결합할 수 있습니다. 동일한 원리를 사용하여 Hi-C 상호 작용 맵을 사용하면 부계 및 모계 자매 염색체에 유전적 변이를 지정하여 이배체 유전체를 생성할 수 있습니다(그림 Hi-C 염기서열 분석 데이터의 다운스트림 응용 분야 참고).