✓ 연중무휴 하루 24시간 자동 온라인 주문 처리
✓ 풍부한 지식과 전문성을 갖춘 제품 및 기술 지원
✓ 신속하고 안정적인 (재)주문
dPCR Microbial DNA Detection Assays
Cat. No. / ID: 250207
✓ 연중무휴 하루 24시간 자동 온라인 주문 처리
✓ 풍부한 지식과 전문성을 갖춘 제품 및 기술 지원
✓ 신속하고 안정적인 (재)주문
특징
- 미생물종, 독성 유전자 또는 항생제 내성 유전자 검출을 위한 사전 설계된 분석법
- 700개 이상의 표적에 대한 분석 항목
- GeneGlobe에서 박테리아, 곰팡이, 바이러스 표적용 맞춤 분석 설계 도구 이용 가능
- 염료 선택을 통해 반응당 최대 5개 타깃까지 멀티플렉싱 가능
- QIAcuity의 간단하고 빠른 dPCR 워크플로우
- QIAcuity OneStep Advanced Probe Kit를 사용하여 한 반응에서 미생물 DNA 및 바이러스 RNA 표적 동시 검출
제품 세부 정보
인간의 건강, 식품 및 사료 테스트, 환경 테스트를 포함한 많은 분야에서 미생물 식별 및 프로파일링에 관심을 두고 있습니다. 미생물 식별은 샘플에서 미생물의 존재 여부를 결정하는 반면 미생물 프로파일링은 두 가지 이상의 실험 조건에서 상대적 발현을 결정하므로 참조 샘플과 노멀라이저가 필요합니다.
당사의 dPCR Microbial DNA Detection Assays는 박테리아, 곰팡이, 기생충, 바이러스, 항생제 내성 또는 독성 인자 유전자를 표적으로 합니다. 박테리아의 경우 분석이 16S rRNA 유전자를 표적으로 하고, 진균의 경우 리보솜 RNA 유전자를 표적으로 합니다. 제품군은 700개 이상의 다양한 분석 항목으로 구성되어 있으며, 그중 200개는 dPCR 웨트 랩(wet-lab) 테스트를 거쳤습니다. 당사는 사전 설계된 미생물 dPCR 분석 제품군에서 다루지 않는 표적을 위해 박테리아, 곰팡이, 바이러스 표적에 대한 간편한 맞춤 분석 설계 도구를 제공합니다.
각 분석은 프라이머 쌍(pair)과 구성 가능한 형광단 염료가 있는 프라이머 프로브로 구성됩니다. 사용 가능한 형광단은 FAM, HEX, ROX, TAMRA 및 Cy5이며, 하나의 멀티플렉스 dPCR 반응에서 최대 5개의 서로 다른 표적 분석 지원을 위해 이들을 혼합 및 일치시킬 수 있습니다. 또한 일반적으로 많이 분석되는 표적들에 대해서는 웨트 랩(wet-lab) 테스트를 거친 dPCR 5-plex 분석 번들을 제공합니다. 자세한 내용은 아래의 애플리케이션을 참조하세요. dPCR 반응의 경우 해당 분석은 QIAcuity Probe PCR Kit(DNA 표적) 또는 QIAcuity OneStep Advanced Probe Kit(RNA 표적)와 함께 사용됩니다.
이 분석은 QIAcuity Digital PCR System 및 QIAcuity Nanoplate와 함께 사용합니다.
이 제품에 대해 자세히 알아보고 당사의 dPCR 전문가에게 문의하시겠습니까? 여기에 정보를 기입하시면 곧 연락드리겠습니다.
성능
정확하고 정밀한 검출
dPCR Microbial DNA Detection Assays 및 QIAcuity dPCR System을 통해 정확한 디지털 PCR 기반 정량화가 가능합니다. NIST 참조 물질의 분석은 입력 템플릿의 예상 농도를 보여줍니다. 자세한 내용은 Shigella sonnei의 gDNA를 사용한 NIST reference standard 8376의 정량화 그림을 참조하세요.
멀티플렉스에서의 성능
dPCR 기반 미생물 표적의 멀티플렉스 검출은 더 적은 수의 반응에서 작은 패널들을 유연하게 설정하게 함으로써 귀중한 샘플을 보존할 수 있습니다. 멀티플렉싱은 또한 더 적은 수의 나노플레이트 웰이 필요하므로 각 실행에서 분석할 수 있는 샘플의 수가 증가함으로써 분석 처리량이 향상됩니다. dPCR Microbial DNA Detection Assays를 이용하여 멀티플렉스 분석을 할 수 있습니다.
단일 분석을 여러 염료(FAM, HEX, TAMRA, ROX 또는 Cy5)와 함께 주문하여 멀티플렉싱을 위해 자체 분석을 결합할 수 있습니다. 또는 당사 과학자들이 이미 dPCR에서 웨트 랩(wet-lab) 테스트를 거친 5-plex 분석 번들 제품을 선택할 수도 있습니다. 위의 번들 제품 외에 700개가 넘는 사전 설계된 분석 항목들의 다른 조합들은 자체 실험실에서 확인해야 합니다.
dPCR Microbial DNA Detection Assays를 사용한 싱글플렉스 및 멀티플렉스 분석 비교는 모든 표적에 대해 유사하고 매우 정밀한 정량화를 보여줍니다(그림 QIAcuity의 멀티플렉스 dPCR에서 미생물 검출 참조). 정밀한 표적 정량화 외에도 5-plex dPCR 실행은 다양한 수중 미생물 병원체의 매우 특이적인 검출을 보여줍니다(그림 QIAcuity의 5-plex를 활용한 수중 미생물 병원체 검출 참조).
원리
사전 설계된 Microbial DNA Detection Assays와 Custom dPCR Microbial Assays는 나노플레이트 디지털 PCR에 사용하기 위한 것입니다. 각 분석은 관련 미생물의 종 특이적 유전 영역 또는 개별 미생물 유전자 영역의 종말점 PCR 증폭을 기반으로 합니다. 증폭된 생성물은 표적 특이적 형광 가수 분해 프로브를 사용하여 검출되며, 이는 분석의 특이성을 향상시킵니다.
박테리아 종 검출을 위한 dPCR Microbial DNA Detection Assays는 RNA 유전자(주로 16S 리보솜 RNA 유전자)를 표적으로 하며, NCBI상의 16S 서열 및 유형 균주 DNA 서열에 대한 GreenGenes 데이터베이스를 사용하여 설계되었습니다. 곰팡이, 바이러스 및 후생동물(metazoan) 종에 대한 분석은 각각 NCBI상의 리보솜 RNA 유전자 및 기타 개별 표지 유전자를 포함하여 다양한 표적 특이적 유전 영역을 표적으로 합니다. 다양한 데이터베이스가 항생제 내성 유전자(예: lahey.org, ARDB 등) 및 독성 인자 유전자(예: VFDB)에 대한 분석 설계에 사용되었습니다.
Custom dPCR Microbial Assays는 미생물 표적을 위해 특별히 개발된 정교하고 철저한 테스트를 거친 알고리즘을 기반으로 하는 맞춤형 분석 설계 소프트웨어로 만들어집니다. 해당 소프트웨어는 각 분석 설계가 엄격한 성능 기준을 충족하여 최적의 민감도와 특이성을 갖춘 강력한 고품질 분석을 제공하도록 보장합니다.
나노플레이트에서의 dPCR 반응 원리는 여기에 설명되어 있습니다.
절차
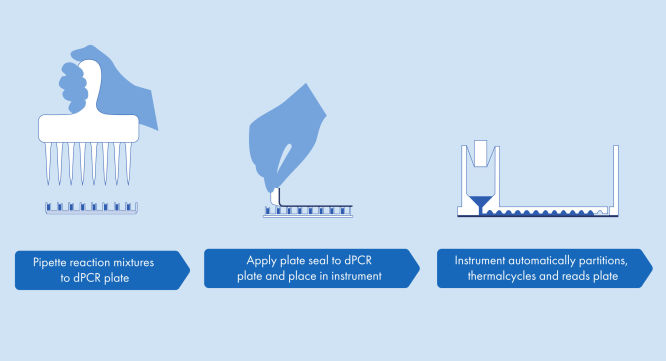
dPCR Microbial DNA Detection Assays 및 Custom dPCR Microbial Assays의 프로토콜은 간단하며 QIAcuity dPCR 기기가 있는 모든 실험실에서 수행할 수 있습니다. 샘플에서 분리된 DNA를 바로 사용할 수 있는 QIAcuity Probe Mastermix 및 Microbial DNA-Free Water(UCP 증류수)에 추가합니다. 이 혼합물을 프라이머 및 가수 분해 프로브 세트가 분주되어 있는 dPCR pre-plate의 각각의 웰에 분주합니다. 반응 혼합물은 pre-plate에서 dPCR 나노플레이트의 웰로 옮겨진 다음 밀봉되어 QIAcuity dPCR 기기로 옮겨집니다(그림 간단하고 신속한 플레이트 기반 워크플로우 참고). 분할, 사이클링 및 이미징 단계는 사전 설정된 매개변수에 따라 QIAcuity dPCR 기기에 의해 완전히 자동화됩니다. 사이클링 프로토콜에 따라 dPCR 실행 결과는 약 2시간 후에 QIAcuity Software Suite에서 분석될 수 있습니다(그림 최소한의 hands-on 시간으로 약 2시간 만에 미생물 dPCR 실험 참조).
응용 분야
dPCR Microbial DNA Detection Assays는 박테리아, 곰팡이 및 바이러스 종과 미생물 항생제 내성 또는 독성 인자 유전자의 검출에 매우 적합합니다. 대변, 가래, 질 면봉(vaginal swab), 하수 등을 포함한 다양한 샘플을 분석할 수 있습니다.
또한 Custom dPCR Microbial 분석 설계 도구를 사용하면 사전 설계된 dPCR Microbial DNA Detection Assays에 포함되지 않은 모든 관심 대상 미생물(박테리아, 곰팡이, 바이러스) 표적에 대한 분석을 원활하게 설계할 수 있습니다.
dPCR 웨트 랩(wet-lab) 테스트를 거친 5-plex 번들
| 번들 ID | 적용 분야 | Assay 카탈로그 번호 및 형광단* | 표적(NCBI 분류 ID) |
| WW-001 | 폐수 1 | DMA00278-F DMA00291-R DMA00340-H DMA00192-T DMA00344-C | Pseudomonas aeruginosa (287) Salmonella enterica (28901) Vibrio cholerae (666) Legionella pneumophila (446) Yersinia enterocolitica (630) |
| WW-002 | 폐수 2 | DMA00340-H DMA00344-R DMA00199-C DMA00192-T DMA00710-F | Vibrio cholerae (666) Yersinia enterocolitica (630) Listeria monocytogenes (1639) Legionella pneumophila (446) Human corona virus SARS-CoV-2 (2697049) |
| WW-003 | 폐수 3 | DMA00109-F DMA00192-H DMA00340-T DMA00194-R DMA00344-C | Clostridium perfringens (1502) Legionella pneumophila (446) Vibrio cholerae (666) Leptospira alexanderi (100053) Yersinia enterocolitica (630) |
| HM-001 | 인간 마이크로바이옴 1 | DMA00148-F DMA00143-T DMA00024-R DMA00003-C DMA00150-H | Faecalibacterium prausnitzii (853) Eubacterium rectale (39491) Akkermansia muciniphila (239935) Acidaminococcus fermentans (951) Finegoldia magna (1260) |
| PB-001 | 프로바이오틱스 1 | DMA00177-F DMA00185-T DMA00061-C DMA00137-R DMA00320-H | Lactobacillus acidophilus (1579) Lactiplantibacillus plantarum (1590) Bifidobacterium bifidum (1681) Enterococcus faecium (1352) Streptococcus salivarius (1304) |
| RG-001 | 저항 유전자 1 | DMA00566-F DMA00542-H DMA00576-T DMA00574-R DMA00575-C | Fluoroquinolone resistance gene QnrS Class D beta-lactamase OXA-10 group Vancomycin resistance gene vanB Tetracycline efflux pump gene tetA Tetracycline efflux pump gene tetB |
| RG-002 | 저항 유전자 2 | DMA00557-T DMA00528-R DMA00548-F DMA00587-H DMA00553-C | Fluoroquinolone resistance geneQepA Class B beta-lactamase blaVIM-1 group Class D beta-lactamase OXA-48 group Sulfonamide resistance gene sul1 (43904) Class D beta-lactamase OXA-58 group |
| VG-001 | 독성 유전자 1 | DMA00614-F DMA00635-T DMA00677-H DMA00680-R DMA00597-C | Minor fimbrial subunit (fimH) Gamma-hemolysin component B (hlgB) Shiga-like toxin 1 subunit B encoded within prophage CP-933V Shiga toxin subunit B; receptor binding subunit Accessory cholera enterotoxin (ace) |
| CP-001 | 칸나비스(Cannabis) 생산 1 | DMA00278-F DMA00302-H DMA00365-R DMA00199-C | Pseudomonas aeruginosa (287) Staphylococcus aureus (1280) Aspergillus niger (5061) Listeria monocytogenes (1639) |
| HM-002 | 인간 마이크로바이옴 2 | DMA00271-F DMA00017-T DMA00049-C DMA00142-H DMA00317-R | Prevotella oralis (28134) Actinomyces viscosus (1656) Bacteroides fragilis (817) Eubacterium infirmum (56774) Streptococcus oralis (1303) |
| RG-003 | 저항 유전자 3 | DMA00579-F DMA00577-T DMA00573-R DMA00502-H DMA00559-C | aac(6')-Ib vanC oprm QnrB-1 group CTX-M-1 Group |
| VG-002 | 독성 유전자 2 | DMA00664-F DMA00677-H DMA00678-T DMA00642-R DMA00680-C | ply stx1B stx2A invA stxB |
| VG-003 | 독성 유전자 3 | DMA00688-F DMA00668-H DMA00596-T DMA00597-R DMA00611-C | wbkA ptxA ace (E. faecalis) ace (V. cholerae) efaA |
* F: FAM, H: HEX, T: TAMRA, R: ROX or C: Cy5
지원되는 데이터 및 수치
간단하고 신속한 플레이트 기반 워크플로우
분획, 서모사이클링 및 이미징이 모두 하나의 완전히 자동화된 기기에 통합되어 두 시간 이내에 결과를 제공합니다. 나노플레이트 형식은 샘플 준비를 위한 프런트 엔드 자동화가 가능하여 직접 실험 시간을 더욱 단축합니다.