✓ 연중무휴 하루 24시간 자동 온라인 주문 처리
✓ 풍부한 지식과 전문성을 갖춘 제품 및 기술 지원
✓ 신속하고 안정적인 (재)주문
E. coli resDNA Quant Standard Kit
Cat. No. / ID: 250221
✓ 연중무휴 하루 24시간 자동 온라인 주문 처리
✓ 풍부한 지식과 전문성을 갖춘 제품 및 기술 지원
✓ 신속하고 안정적인 (재)주문
특징
- 간편한 설정 및 숙주 세포 DNA 검출을 위한 대조물질이 포함된 사전 혼합된 마스터 혼합물
- 대장균(Escherichia coli, E.coli), 중국 햄스터 난소(Chinese Hamster Ovary, CHO), 인간 배아 신장 293(Human Embryonic Kidney 293, HEK293) 세포에서 적게는 펨토그램 수준까지 잔류 숙주 세포 DNA를 정확하게 검출할 수 있음
- 고도로 단편화된 숙주 세포 DNA를 검출하고 정량화하는 멀티카피 표적 분석
- 추출된 샘플과 추출되지 않은 샘플 모두에서 숙주 세포 DNA 검출 가능
제품 세부 정보
백신, 약물 및 기타 바이오 의약품과 같은 제품의 숙주 세포 DNA 오염은 건강에 큰 위험을 초래합니다. 따라서 미국 FDA 및 WHO와 같은 기관에서 안전 한도를 엄격하게 규제하고 있습니다. 약물 투여의 특성, 감염성, 오염 세포 DNA의 발암성에 따라 잔류 DNA 상한에 대한 명확한 지침이 설정됩니다. 예를 들어, 비종양원성 세포 DNA 비경구 투여 시 1회 투여량당 10ng, 최대 길이 200bp로 제한해야 하며, 경구 투여 백신의 경우 잔류 DNA는 1회 투여량당 100µg 미만으로 제한하는 것이 WHO의 권장 사항입니다.
제조 제품에서 이러한 오염을 검출하고 제거하려면 제품에 존재하는 극미량의 특정 숙주 세포 DNA를 매우 민감하고 정확하게 측정해야 합니다.
디지털 PCR은 잔류 DNA 정량화를 위해 선호되는 검출 방법입니다. 이는 특히 qPCR과 같은 다른 검출 방법과 비교하여 미량의 오염 검출에서 뛰어난 민감도와 정확성을 제공하기 때문입니다. QIAcuity Residual DNA Quantification Kits는 대장균(Escherichia coli, E.coli), 중국 햄스터 난소(Chinese Hamster Ovary, CHO) 세포, 인간 배아 신장 293(Human Embryonic Kidney 293, HEK293) 세포 숙주 세포 DNA를 정확하고 정밀하게 검출할 수 있습니다.
성능
QIAcuity Residual DNA Quantification Kits는 PCR 오염 물질 및 기타 억제 시약이 있는 경우에도 추출 여부와 관계없이 중국 햄스터 난소(Chinese Hamster Ovary, CHO), 대장균(Escherichia coli, E. coli), 인간 배아 신장 293(Human Embryonic Kidney 293, HEK293)의 정확한 잔류 DNA(residual DNA, resDNA) 정량화 결과를 제공합니다. 이 분석은 고도로 단편화된 잔류 숙주 세포 DNA를 정확하게 측정하는 멀티카피 표적 분석입니다.
| 분석 | 표적 복제수 | 앰플리콘 크기 | 변환 계수 cp/µl~fg/µl |
|---|---|---|---|
| QIAcuity E.coli resDNA Quant Kit | 7 | <200 | 0.35 |
| QIAcuity CHO resDNA Quant Kit | 약 1백만, 정의되지 않음(반복 요소) | <100 | 0.28 |
| QIAcuity HEK293 resDNA Quant Kit | 약 1백만, 정의되지 않음(반복 요소) | <100 | 1.54 |
QIAcuity E.coli resDNA Quant Kit는 단일 반응에서 적게는 5fg까지 잔류 DNA를 검출합니다
| 반응당 로딩양(fg/rxn) | 대장균(Escherichia coli, E.coli) 표준액(copies/µl) | 내부 대조물질(copies/µl)* |
|---|---|---|
| 50000 | 2949.7 | 99.9 |
| 5000 | 291.6 | 91.7 |
| 500 | 27.6 | 91.4 |
| 50 | 2.8 | 93.2 |
| 25 | 1.46 | 92.5 |
| 5 | 0.45 | 90.6 |
| NTC | 0 | 98.9 |
*예상되는 내부 대조물질: 100copies/µl
디지털 PCR은 qPCR에 비해 더 낮은 템플릿 농도 범위에서 더 높은 검출 민감도를 제공하므로 더 견고한 애플리케이션 적용이 가능합니다.
QIAcuity CHO resDNA Quant Kit는 단일 반응에서 적게는 5fg까지의 잔류 DNA를 검출합니다
| 반응당 로딩양(fg/rxn) | 중국 햄스터 난소(Chinese Hamster Ovary, CHO) 표준액(copies/µl) | 내부 대조물질(copies/µl)* |
|---|---|---|
| 50000 | 4939.3 | 108.7 |
| 5000 | 508.3 | 104.9 |
| 500 | 50.2 | 97.6 |
| 50 | 4.7 | 99.5 |
| 25 | 2.6 | 101.3 |
| 5 | 0.6 | 100.4 |
| NTC | 0 | 104.7 |
*예상되는 내부 대조물질: 100copies/µl
QIAcuity HEK293 resDNA Quant Kit는 단일 반응에서 적게는 5fg까지의 잔류 DNA를 검출합니다
| 반응당 로딩양(fg/rxn) | 인간 배아 신장 293(Human Embryonic Kidney 293, HEK293) 표준액(copies/µl) | 내부 대조물질(copies/µl)* |
|---|---|---|
| 50000 | 1104.9 | 96.6 |
| 5000 | 104.5 | 92.5 |
| 500 | 8.83 | 92.4 |
| 50 | 0.99 | 94.4 |
| NTC | 0 | 96.2 |
*예상되는 내부 대조물질: 100copies/µl
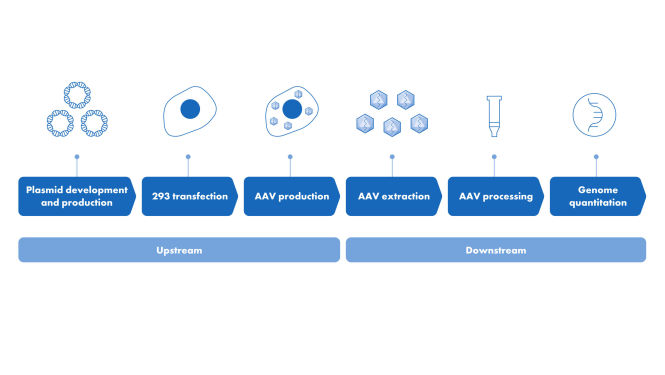
이 키트는 QIAcuity Digital PCR System 및 QIAcuity Nanoplate와 함께 사용하며 qPCR에 필적하는 엔드 투 엔드 dPCR 작업 흐름을 제공하는 반면 시료에서 잔류 DNA(residual DNA, resDNA)의 절대 정량화를 제공합니다. 키트는 바이오프로세스 제조 및 품질 관리(Quality Control, QC) 요구 사항을 염두에 두고 설계되었습니다.
원리
나노플레이트에서의 dPCR 반응 원리는 여기에 설명되어 있습니다.
숙주 세포 DNA 모니터링을 위해 QIAcuity 디지털 PCR을 사용하면 QIAcuity Nanoplate 26k를 통해 벌크 샘플의 파티셔닝으로 검출 한계(Limit of Detection, LOD)/정량 한계(Limit of Quantitation, LOQ)가 향상됩니다. 파티셔닝은 표적의 유효 농도를 늘려 소량의 숙주 세포 DNA를 보다 정확하고 정밀하게 캡처하고 측정할 수 있도록 합니다. 더 많은 파티션 수를 가진 QIAcuity Nanoplate 26k에 더 많은 템플릿을 로딩할 수 있다는 점이 잔류 숙주 세포 검출의 정확도와 정밀도를 더 높이는 이유 중 하나입니다.
절차
Lysate를 residual quant assay와 내부 대조 물질(IC)과 함께 마스터 믹스에 추가한 후 숙주 세포 DNA 정량을 수행합니다. Copies/µl은 제공된 변환 계수를 통해 fg/µl로 변환됩니다.
응용 분야
QIAcuity Residual DNA Quantification Kits는 복잡한 바이오프로세스 중간체에서 숙주 세포 DNA를 정밀하게 정량화하는 데 이상적입니다.
지원되는 데이터 및 수치
최종 물질에는 제한된 수준의 불순물만 허용됩니다
숙주 세포 DNA의 캐리오버는 안전 문제를 초래하며 FDA 및 WHO와 같은 당국에 의해 엄격하게 규제됩니다