✓ 연중무휴 하루 24시간 자동 온라인 주문 처리
✓ 풍부한 지식과 전문성을 갖춘 제품 및 기술 지원
✓ 신속하고 안정적인 (재)주문
QuantiTect Virus Kit (1000)
Cat. No. / ID: 211015
✓ 연중무휴 하루 24시간 자동 온라인 주문 처리
✓ 풍부한 지식과 전문성을 갖춘 제품 및 기술 지원
✓ 신속하고 안정적인 (재)주문
특징
- 싱글 및 멀티플렉스 분석에서의 높은 민감도
- 동일한 반응에서 바이러스 RNA 및/또는 DNA 검출
- 약한 양성 신호의 명확한 검출
- 신속한 범용 투스텝 프로토콜
- 더 많은 샘플 투입이 가능한 더 뛰어난 민감도의 5x 마스터 믹스
제품 세부 정보
성능
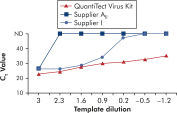
QuantiTect Virus Kits를 사용한 증폭은 높은 CT 값의 적은 템플릿 양으로도 다양한 희석 범위에 걸쳐 가파른 시그모이드 곡선을 제공합니다(그림 ' 넓은 동적 범위에서 명확한 CT 값 결정' 참고). 이로써 real-time PCR에서 바이러스 핵산의 정량화를 위한 정확한 CT 값을 결정할 수 있습니다.
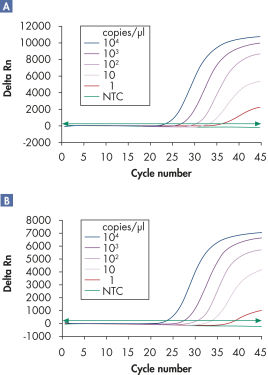
멀티플렉스 분석은 민감도 손실 없이 넓은 선형 범위에서 여러 바이러스 RNA 및/또는 DNA 표적과 내부 대조물질을 검출할 수 있습니다(그림 ' 넓은 선형 범위에서 바이러스 RNA의 안정적인 검출' 및 ' 향상된 소량의 바이러스 RNA 검출 능력' 참고).
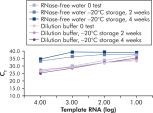
키트와 함께 제공되는 QuantiTect 핵산 희석 완충액은 희석 및 반응 셋업 중에 RNA 및 DNA 표준물질을 안정화시키고 튜브나 피펫 팁과 같은 플라스틱 표면에서 핵산의 손실을 방지합니다. 이 완충액은 바이러스 핵산을 정량화하는 데 사용되는 표준물질을 안정적으로 희석하여 낮은 CT 값에서 높은 CT 값까지 넓은 선형 범위를 제공하고, 표준물질을 품질 저하 없이 오래 보관할 수 있도록 해줍니다(그림 ' RNA 표준물질의 안정적인 희석 및 보관' 참고).
그림 참조
원리
QuantiTect Virus Kits는 한 번의 시도로 싱글 또는 멀티플렉스 분석에서 고감도 바이러스 핵산 검출을 제공합니다(순서도 ' QIAGEN 멀티플렉스 키트' 참고). 최적화된 마스터 믹스는 멀티플렉스 반응의 PCR 생성물이 대응하는 단일 증폭 반응의 PCR 산물과 동일한 효율과 민감도로 증폭되도록 보장합니다.
대조물질과 표적 유전자를 별도의 반응이 아닌 동일한 반응에서 증폭하면 취급 오류를 최소화하여 유전자 정량화의 신뢰성을 높일 수 있습니다. QuantiTect Virus 완충액에는 K+ 및 NH4+ 이온의 균형 잡힌 조성과 독자적인 합성 인자 MP 안정제가 함유되어 있어 프라이머와 프로브를 핵산 템플릿에 안정적이고 효율적으로 어닐링하여 높은 PCR 효율을 가능하게 합니다(그림 독자적인 PCR 완충액 참고). 또한 Sensiscript 역전사 효소의 독자적인 배합으로 바이러스 RNA의 고감도 역전사를 보장하며, HotStarTaq Plus DNA Polymerase는 엄격한 hot start를 제공하여 비특이적 생성물의 형성을 방지합니다.
| 키트 구성품 | 특징 | 이점 | |
|---|---|---|---|
| 5x QuantiTect Virus 마스터 믹스 | 농축 마스터 믹스 | 고감도 바이러스 검출에 최적화된 고농축 제품 | 민감도 향상을 위해 더 많은 양의 템플릿을 분석에 추가할 수 있음 |
| HotStarTaq Plus DNA Polymerase | 95ºC 5분 활성화 | 실온에서 qPCR 반응 셋업 | |
| QuantiTect Virus 완충액 | NH4+와 K+ 이온의 균형 잡힌 조성 | 특이적 프라이머 어닐링으로 신뢰할 수 있는 PCR 결과 보장 | |
| 합성 인자 MP | 같은 튜브에서 최대 4개의 유전자를 안정적으로 멀티플렉싱 분석 | ||
| 추가 키트 구성품 | QuantiTect Virus RT 혼합물 | Sensiscript 역전사 효소의 독자적인 배합 함유 | 바이러스 RNA의 고감도 검출에 최적화됨 |
| QuantiTect 핵산 희석 완충액 | 핵산 표준물질의 희석 및 보관을 위한 독점적 배합의 완충액 | 희석 및 반응 셋업 중에 RNA 및 DNA 표준물질을 안정화시키고 튜브나 피펫 팁과 같은 플라스틱 표면에서 핵산의 손실을 방지 |
그림 참조
절차
QuantiTect Virus Kits는 서열 특이적 프로브를 사용하여 바이러스 핵산(RNA 및/또는 DNA)의 고감도 real-time PCR 분석 및 내부 대조물질을 제공합니다. 역전사 단계의 유무와 관계없이 반응을 수행할 수 있으므로 RNA 표적, DNA 표적 또는 RNA와 DNA 표적을 모두 검출하기 위한 멀티플렉스 분석을 유연하게 설계할 수 있습니다. 빠르고 안정적인 결과를 얻으려면 안내서의 프로토콜을 따르십시오.
마스터 믹스 내 ROX 패시브 기준 염료의 유무와 관계없이 키트를 사용할 수 있습니다(표 참고).
| ROX 염료 | 키트 | 호환되는 사이클러 |
|---|---|---|
| 마스터 믹스 내에 제공됨 | QuantiTect Virus Kit | Applied Biosystems 7500을 제외한 Applied Biosystems의 모든 사이클러 |
| 별도의 튜브로 제공 | QuantiTect Virus +ROX Vial Kit | Applied Biosystems 7500 및 Bio-Rad, Cepheid, Eppendorf, QIAGEN, Roche, Agilent 및 기타 공급업체의 사이클러 |
바이러스 검출을 포함한 신속하고 높은 민감도의 end-point 원스텝 RT-PCR 응용 분야에서는 QIAGEN OneStep RT-PCR Kit 사용을 권장합니다.
응용 분야
지원되는 데이터 및 수치
넓은 동적 범위에서 명확한 CT 값 결정.
Specifications
| Features | Specifications |
|---|---|
| Applications | 바이러스 검출 |
| SYBR Green I or sequence-specific probes | 서열 특이적 프로브 |
| Real-time or endpoint | Real-Time |
| Reaction type | 역전사 및 PCR |
| Thermal cycler | 대부분의 real-time 사이클러( LightCycler® 1.x 및 2.0과 같은 capillary 사이클러 제외) |
| Sample/target type | RNA 및/또는 DNA 표적 |
| With or without ROX | 마스터 믹스에 ROX가 포함되거나 ROX가 별도 바이알로 제공 |
| Single or multiplex | 싱글 또는 멀티플렉스 |