✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
dPCR Microbial DNA Detection Assays
Cat. No. / ID: 250207
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特徴
- 微生物種、病原性因子遺伝子、ウイルス、抗生物質耐性遺伝子を検出
- 680以上のターゲットをカバーするアッセイ
- 1回の反応で最大5種類のターゲットのマルチプレックス検出が可能な色素をラインナップ
- QIAcuityでシンプルかつ迅速なdPCRワークフローを実現
- QIAcuity OneStep Advanced Probe Kitを使用することで、1回の反応で微生物DNAとウイルスRNAの両方をターゲットに設定
製品詳細
微生物同定およびプロファイリングは、ヒト健康、食品および飼料検査、環境検査など多くの分野で関心を集めています。微生物同定は、サンプル中の微生物の有無を決定するのに対して、微生物プロファイリングは、2つ以上の実験条件下で相対的発現を決定することから、リファレンスサンプルとノーマライザーを必要とします。
当社のdPCR Microbial DNA Detection Assaysは、細菌、真菌、寄生虫、ウイルス、抗生物質耐性や病原性因子遺伝子をターゲットとします。細菌は16S rRNA遺伝子を、真菌はリボソームRNA遺伝子をターゲットとします。680種類以上のアッセイで構成されるポートフォリオを提供し、そのうち200種類はウェットラボでのdPCR検証済みです(アッセイの詳細は GeneGlobeをご参照ください)。
各アッセイは、プライマーペアおよびユーザーが構成可能な蛍光色素を使用した加水分解プローブで構成されます。蛍光色素はFAM、HEX、ROX、TAMRAおよびCy5を使用可能です。これらの色素を組み合わせることで、1回のマルチプレックスdPCR反応で最大5種類のターゲット解析が可能になります。一般的に関心の高いターゲットの解析に対応した、ウェットラボで検証済みの5プレックスdPCRアッセイセットも提供しています。詳細については以下をご参照ください。dPCR反応には、本アッセイと合わせて QIAcuity Probe PCR Kit (DNAターゲット)またはQIAcuity OneStep Advanced Probe Kit(RNAターゲット)をお使いください。
このキットは、QIAcuity Digital PCR System およびQIAcuity Nanoplatesと併用してお使いいただけます。
この製品について詳細をご希望、または、当社のdPCRの担当者より連絡をご希望の場合、こちらからサインインしていただくと、すぐに当社からご連絡を差し上げます。
パフォーマンス
正確かつ精密な検出
dPCR Microbial DNA Detection AssaysおよびQIAcuity dPCR Systemを使用することで、正確なdPCR定量が可能です。NIST標準物質の解析の結果、予想どおりの入力テンプレート濃度が得られました。詳細は、図 Shigella sonneiのgDNAを用いたNIST標準物質8376の定量 をご参照ください
マルチプレックス性能
微生物ターゲットの検出にマルチプレックスdPCRを用いることで、小型パネルの柔軟なセットアップとより少ない反応回数での解析が可能になり、貴重なサンプルを節約できます。また、マルチプレックス化によって、必要なナノプレートのウェル数が少なくて済みます。これによって、解析スループットが向上し、1回のランでより多くのサンプルを解析できます。dPCR Microbial DNA Detection Assaysは、マルチプレックス解析に組み込み可能です。
さまざまな色素(FAM、HEX、TAMRA、ROX、Cy5)のシングルアッセイを注文することで、独自のマルチプレックスアッセイをつくることができます。あるいは、ウェットラボでdPCR検証済みの5プレックスアッセイセットからお選びいただくこともできます。その他685種類以上のアッセイの組み合わせは、お客様のラボでの検証が必要です。
dPCR Microbial DNA Detection Assaysによるシングルプレックス解析およびマルチプレックス解析を比較した結果、同様にすべてのターゲットを精密に定量できました(図QIAcuityでのマルチプレックスdPCRによる微生物検出を参照)。5プレックスdPCR解析では、精密なターゲット定量に加えて、さまざまな水中病原性微生物を非常に特異的に検出できました(図QIAcuityでの5プレックス解析による水中病原性微生物の検出を参照)。
原理
dPCR Microbial DNA Detection AssaysはナノプレートdPCRでの使用を目的としています。各アッセイは、関連する微生物の種特異的な遺伝子領域、 または個々の微生物の遺伝子領域に対して、エンドポイントでPCR増幅を行います。ターゲット特異的な蛍光加水分解プローブで増幅産物を検出することによって、アッセイの特異性を高めます。
細菌種検出アッセイは、リボソームRNA遺伝子(主に16SリボソームRNA遺伝子)をターゲットとし、NCBIが管理する16Sシークエンスおよび基準株DNAシークエンスのGreenGenesデータベースをもとに設計されています。真菌、ウイルスおよび後生動物種向けアッセイは、NCBIが管理するリボソームRNA遺伝子やその他マーカー遺伝子など、ターゲットに特異的な遺伝子領域をターゲットとします。抗生物質耐性遺伝子(lahey.org, ARDBなど)および病原性因子遺伝子(virulence factor genes、VFDB)向けアッセイは、各種データベースをもとに設計されています。
ナノプレートでのdPCR反応の原理はこちらで説明しています。
操作手順
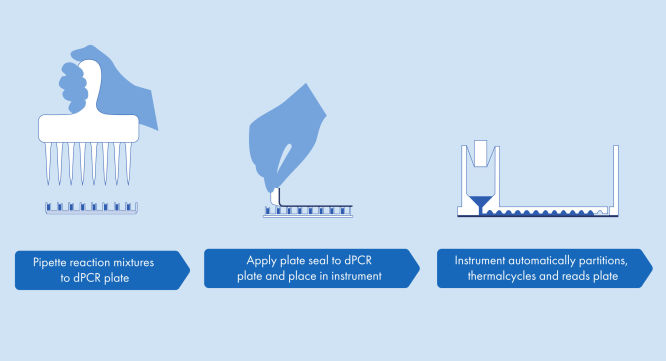
dPCR Microbial DNA Detection Assayはシンプルなプロトコールを採用しており、QIAcuity dPCR装置があるラボならどこでも使用できます。サンプルからDNAを単離して、そのまま使えるQIAcuity Probe MastermixとMicrobial DNA-Free Water(UCP水)に添加します。この混合液を、プライマーと加水分解プローブがあらかじめ注入されたdPCRプリプレートの各ウェルに分注します。反応液をプリプレートからdPCR Nanoplateのウェルに移し、密封してQIAcuity dPCR装置に移します(図プレートを使用したシンプルで迅速なワークフロー)を参照)。QIAcuity dPCR装置があらかじめ設定されたパラメーターに従ってパーティショニング、サイクリング、およびイメージングを全自動処理します。サイクリングプロトコールに応じて、2時間以内にQIAcuity Software SuiteでdPCRランの結果を解析できます(図最小限の作業時間の約2時間でdPCR微生物解析を実現を参照)。
アプリケーション
dPCR Microbial DNA Detection Assaysは、細菌、真菌、ウイルス種、抗生物質耐性および病原性因子遺伝子の検出に非常に適しています。糞便、唾液、膣スワブ、下水、その他幅広いサンプルの解析が可能です。
ウェットラボでの5プレックスdPCR検証済みです。
| Bundle ID | Application Field | Assay Catalog Numbers and Fluorophore* |
Targets (NCBI Taxonomy ID) |
| WW-001 | Wastewater 1 | DMA00278-F DMA00291-R DMA00340-H DMA00192-T DMA00344-C |
Pseudomonas aeruginosa (287) Salmonella enterica (28901) Vibrio cholerae (666) Legionella pneumophila (446) Yersinia enterocolitica (630) |
| WW-002 | Wastewater 2 | DMA00340-H DMA00344-R DMA00199-C DMA00192-T DMA00710-F |
Vibrio cholerae (666) Yersinia enterocolitica (630) Listeria monocytogenes (1639) Legionella pneumophila (446) Human corona virus SARS-CoV-2 (2697049) |
| WW-003 | Wastewater 3 | DMA00109-F DMA00192-H DMA00340-T DMA00194-R DMA00344-C |
Clostridium perfringens (1502) Legionella pneumophila (446) Vibrio cholerae (666) Leptospira alexanderi (100053) Yersinia enterocolitica (630) |
| HM-001 | Human Microbiome 1 | DMA00148-F DMA00143-T DMA00024-R DMA00003-C DMA00150-H |
Faecalibacterium prausnitzii (853) Eubacterium rectale (39491) Akkermansia muciniphila (239935) Acidaminococcus fermentans (951) Finegoldia magna (1260) |
| PB-001 | Probiotics 1 | DMA00177-F DMA00185-T DMA00061-C DMA00137-R DMA00320-H |
Lactobacillus acidophilus (1579) Lactiplantibacillus plantarum (1590) Bifidobacterium bifidum (1681) Enterococcus faecium (1352) Streptococcus salivarius (1304) |
| RG-001 | Resistance Genes 1 | DMA00566-F DMA00542-H DMA00576-T DMA00574-R DMA00575-C |
Fluoroquinolone resistance gene QnrS Class D beta-lactamase OXA-10 group Vancomycin resistance gene vanB Tetracycline efflux pump gene tetA Tetracycline efflux pump gene tetB |
| RG-002 | Resistance Genes 2 | DMA00557-T DMA00528-R DMA00548-F DMA00587-H DMA00553-C |
Fluoroquinolone resistance geneQepA Class B beta-lactamase blaVIM-1 group Class D beta-lactamase OXA-48 group Sulfonamide resistance gene sul1 (43904) Class D beta-lactamase OXA-58 group |
| VG-001 | Virulence Genes 1 | DMA00614-F DMA00635-T DMA00677-H DMA00680-R DMA00597-C |
Minor fimbrial subunit (fimH) Gamma-hemolysin component B (hlgB) Shiga-like toxin 1 subunit B encoded within prophage CP-933V Shiga toxin subunit B; receptor binding subunit Accessory cholera enterotoxin (ace) |
| CP-001 | Cannabis Production 1 | DMA00278-F DMA00302-H DMA00365-R DMA00199-C |
Pseudomonas aeruginosa (287) Staphylococcus aureus (1280) Aspergillus niger (5061) Listeria monocytogenes (1639) |
| HM-002 | Human Microbiome 2 | DMA00271-F DMA00017-T DMA00049-C DMA00142-H DMA00317-R |
Prevotella oralis (28134) Actinomyces viscosus (1656) Bacteroides fragilis (817) Eubacterium infirmum (56774) Streptococcus oralis (1303) |
| RG-003 | Resistance Genes 3 | DMA00579-F DMA00577-T DMA00573-R DMA00502-H DMA00559-C |
aac(6')-Ib vanC oprm QnrB-1 group CTX-M-1 Group |
| VG-002 | Virulence Genes 2 | DMA00664-F DMA00677-H DMA00678-T DMA00642-R DMA00680-C |
ply stx1B stx2A invA stxB |
| VG-003 | Virulence Genes 3 | DMA00688-F DMA00668-H DMA00596-T DMA00597-R DMA00611-C |
wbkA ptxA ace (E. faecalis) ace (V. cholerae) efaA |
* F: FAM, H: HEX, T: TAMRA, R: ROX or C: Cy5
裏付けデータと数値
プレート方式のシンプルで迅速なワークフロー
パーティショニング、サーマルサイクル、イメージングを1つに統合した完全自動化装置が2時間未満で結果を提供します。ナノプレート方式はフロントエンドのサンプル調製の自動化に適しており、手作業時間をさらに短縮できます。