RT2 PreAMP Pathway Primer Mix
Cat. No. / ID: 330241
特徴
- ヒト、マウス、ラット用プライマーミックス
- 最小限の手作業時間
- 少量サンプルで適応性の高いパフォーマンス
製品詳細
RT² PreAMP cDNA Synthesis KitおよびRT² PreAMP Pathway Primer Mixesは、わずか1 ngのトータルRNAからの遺伝子発現解析を実現する画期的なテクノロジーです。独自の増幅プロセスにより、PCRアレイ解析に使用するためのcDNA 量を増やします。スタートサンプルの種類には、細針生検、LMD(レーザーマイクロダイセクション)サンプル、幹細胞クラスターまたは胚様体、FACS(蛍光励起細胞選出法)で分離した細胞集団が含まれます。ヒト、マウス、ラットのRT2 Profiler PCR Arrayの全カタログ商品に対応したプライマーミックスが入手可能です。
パフォーマンス
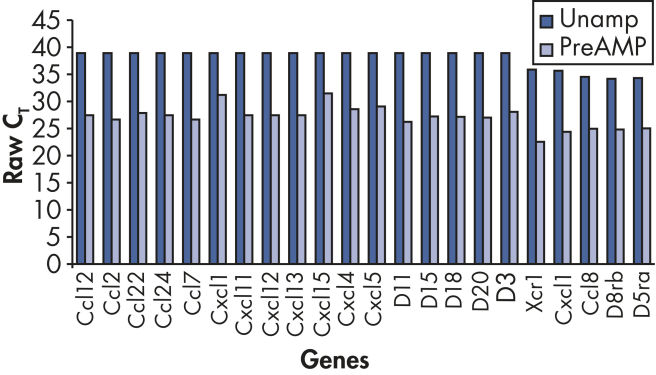
ポジティブコールレートの増加とバイアスのない増幅で信頼性の高い結果
RT² PreAMP Pathway Primer Mixを用いた事前増幅により、さらに多くの遺伝子検出が可能になります(図 “ ポジティブコールレートの増加”)。
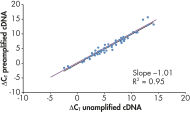
増幅プロセスはバイアスがないので、事前増幅したcDNAと増幅していないcDNAのΔCT値は非常に類似した結果が得られ、これは回帰分析で検証されます(図 “ バイアスのない増幅プロセス”)。
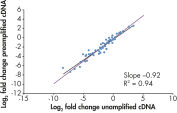
事前増幅したFFPEサンプルと事前増幅していないサンプルの間で、遺伝子発現のfold changeに高い相関性があります(図“ 生体内の発現状態をそのまま増幅”)。
図参照
原理
図参照
操作手順
まずRT2 PreAMP cDNA Synthesis Kitを用いて最高12種類のRNAサンプルから一本鎖cDNAを合成します。その後パスウエイに特異的な遺伝子セットの事前増幅を行ないます。各一本鎖cDNA合成反応液は最高4種類のRT2 PreAMP Pathway Primer Mixesを用いて増幅でき、最高4種類のRT2 Profiler PCR Arrayで遺伝子発現解析を行なえます。RT2 PreAMP cDNA Synthesis Kitに付属のSide Reaction Reducerは事前増幅からの残存プライマーを減らすことで、RT2 Profiler PCR Arrayでの正確な検出を実現します。事前増幅したテンプレートと装置に特異的で即使用可能なRT² SYBR® Green Mastermixを混和すると、PCRアレイ操作は完了します。
アプリケーション
裏付けデータと数値
Increased positive call rate.