QIAGEN Multiplex PCR Kit
至適化なしに特異性および感度の高いマルチプレックスPCR
至適化なしに特異性および感度の高いマルチプレックスPCR
Cat. No. / ID: 206143
QIAGEN Multiplex PCR Kitは即使用可能なマスターミックス・フォーマットとしてお届けします。QIAGEN Multiplex PCR Kit には、HotStarTaq DNA Polymerase の入ったQIAGEN Multiplex PCR Master Mix、およびFactor MP を含むPCR バッファーが添付されています。至適化済みの塩濃度とともに、Factor MP は特異的に結合したプライマーを安定化し、至適化なしに反応液中の全プライマーで効率的なエクステンションを実現します。増幅困難なテンプレート(GC リッチ)用に画期的なQ-Solution も添付されています。
QIAGEN Multiplex PCR Kitは他社に比べて高性能で、特異性と感度の高いマルチプレックス増幅を実現します(図“ 16plex PCR成功例 ”)。本キットはトランスジェニック生物のタイピング(図 " トランスジェニック・マウスのジェノタイピング")やマイクロサテライト解析(図 " マイクロサテライト解析成功例 ") などの様々なタイプのアプリケーションに最適です。マスターミックス中のHotStarTaq DNA Polymeraseは複数のターゲットを同時に効率的に増幅します。マスターミックスに入っている画期的なPCRバッファーにより、増幅効率はさらに改善されています。ユニークなバッファーは、最適化の必要性を最小限に抑え、広範のPCR条件で特異性の高いPCRを実証します。GCリッチなテンプレートを増幅するために添加されているQ - Solution(キットに付属)により、不適切なPCR条件を改善することができます。
濃度:5 units/µl
組み換え酵素:Yes
基質アナログ:dNTP、ddNTP、dUTP、biotin-11-dUTP、DIG-11-dUTP、 fluorescent-dNTP/ddNTP
エクステンション速度:72℃ で2~4 kb/min
半減期:97℃で10分、94℃で60分
増幅効率:≥105 倍
5'–>3' エキソヌクレアーゼ活性:Yes
余分なA付加:Yes
3'–>5' エキソヌクレアーゼ活性:No
ヌクレアーゼの混入:No
RNaseの混入:No
プロテアーゼの混入:No
自己プライマー活性:No
QIAGEN Multiplex PCR Kit はマルチプレックスPCR用に特別に開発された初めてのキットで、使い易いマスターミックスフォーマットが添付されています。QIAGEN Multiplex PCR Master Mixには前もって至適化された濃度のHotStarTaq DNA Polymerase、MgCl2、dNTPsおよびマルチプレックスPCR反応用に特に開発したPCRバッファーが含まれています。本キットでは最初のマルチプレックスPCR実験で良好な結果を実現します。以下のような最適化済みの試薬や酵素により、本キットでは反応条件(例;プライマー、Mg2+、Taq DNA polymeraseの濃度)およびサイクリングパラメーターの至適化は不要です。
HotStarTaq DNA PolymeraseはTaqDNA polymeraseを化学的に修飾したもので、常温では活性がありません。これにより、低温でのPCRセットアップや最初のPCRサイクルで非特異的な増幅産物およびプライマーダイマーの形成を防ぎます。HotStarTaq DNA Polymeraseは、95℃、15 分間のインキュベーションステップで活性化され、このステップは既存のサーマルサイクリングのプログラムに容易に導入できます。
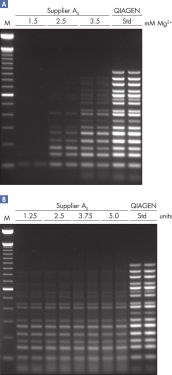
この特殊なバッファーは最適な配合比のK+とNH4+、ならびに革新的なPCR添加物であるFactor MPが入っています。 これはテンプレート周辺でのプライマー濃度を高めます。K+ や他の陽イオンと共に、Factor MPは特異的に結合したプライマーを安定化し、HotStarTaq Plus DNA Polymerase による効果的なプライマー・エクステンションを可能にします(図“ 安定で効率的なプライマー・アニーリング”)。画期的なバッファーは、各PCR ステップのアニーリングステップ中でプライマーの特異的結合の割合を非特異的結合に対して高めることにより、各PCRサイクルの特異的な増幅を実現します。このバッファーはユニークな配合比のKClと (NH4)2SO4を含み、従来のPCRバッファーに比べ、幅広いアニーリング温度やMg2+濃度の範囲で厳密で特異的なプライマー・アニーリングを実現します。従って、異なるアニーリング温度あるいはMg2+ 濃度を用いて行なうPCRの至適化は最小限ですみ、または不要なこともあります。
HotStarTaq DNA Polymeraseに添付されている画期的なPCR添加剤であるQ-Solution は、DNA の変性環境を改善し、増幅困難なテンプレートの増幅を可能にします。このユニークな試薬により、高度な二次構造をもつテンプレートやGC リッチなテンプレートなどにより生じる不適切なPCR 条件が改善されることがあります。Q-Solutionは、DMSOのような通常使用される他のPCR添加物とは異なり、毒性がなく、一定の濃度で使用でき、PCRの正確性を損なわずにPCR純度を確保します。
QIAGEN Multiplex PCR Kit にはすでに至適化され即使用可能なマスターミックスが添付され、さらに使いやすくなっています。マスターミックスを使用すると、時間の節約、反応セットアップの簡略化、ピペッティングエラーやコンタミの発生源排除による再現性向上を実現します - ピペッティング操作は最小限に抑えられ、時間のかかる計算は不要です。プライマートテンプレートのみを添加して最終の増幅ミックスを調製します。マスターミックスは2~8℃で保存できるので、マルチプレックスPCRアッセイのセットアップをさらに迅速に行なえます。キットに付属の詳細なプロトコールは、迅速で容易なPCRセットアップを実現します。反応は室温で設定することができるので、使いやすく便利です。HotStarTaq DNA Polymeraseは、95℃、15 分間のインキュベーションステップで活性化され、このステップは既存のサーマルサイクリングのプログラムに容易に導入できます。
QIAGEN Multiplex PCR Kitは様々なタイプのアプリケーションに最適です。
| Features | Specifications |
|---|---|
| Applications | PCR, RT-PCR, multiplex PCR, typing, detection |
| Enzyme activity | 5' -> 3' exonuclease activity |
| Reaction type | PCR amplification |
| With/without hotstart | With hotstart |
| Single or multiplex | Multiplex |
| Real-time or endpoint | Endpoint |
| Mastermix | Yes |
| Sample/target type | Genomic DNA and cDNA |