QuantiNova Probe PCR Kit (100)
Cat. No. / ID: 208252
特徴
- 内蔵の視覚的インジケーターを用いる正確な反応セットアップ
- 1コピーまでの標的を正確かつ堅牢に検出
- 新しいホットスタートメカニズムによる比類のない特異性
- 1本のチューブで最大5つの標的を高感度検出
- 柔軟なワークフローに対応する30°Cで最大100時間の反応安定性
製品詳細
特異性、感度、速度、プロセス安全性が強化されたQuantiNova SYBR Green PCR Kit(SYBR qPCRキット)は、SYBR Greenベースの qPCRに新たなスタンダードを定めました。新しいホットスタートメカニズムにより、real-time PCRの特異性が大幅に向上し、最適化されたケミストリーによって、あらゆるリアルタイムPCRサイクラーで、cDNAまたはgDNAから正確な定量結果が得られ、ひとつの標的コピーであっても検出できます。最後に、内蔵の視覚的インジケーターにより、正確なピペッティングが保証されます。
QuantiNova Probe PCR Kit(プローブqPCRキット)を使用すると、低温でのミスプライミングによる有害な影響を回避できます。新しい抗体を介したホットスタートメカニズムに基づき、このキットはプローブベースのreal-time PCRの特異性と効率を高め、さまざまなreal-time PCRサイクラーで、正確なシングルプレックスまたはデュプレックスのcDNAまたはgDNA分析を提供します。冷却剤を必要とせず、室温で最大100時間、きわめて安定しているという利便性も備えているため、ハイスループットサンプルの取り扱いや自動ワークフローに理想的です。正確なピペッティングを視覚的に識別するトラッキングシステムを内蔵しているため、絶対的な安心感を得ることができる一方、標的量が少なくても非常に高感度で検出できるため、毎回、信頼性の高いqPCR結果を得ることができます。
QuantiNova Multiplex PCR Kits(マルチプレックスqPCRキット)を使用すると、マルチプレックスreal-time PCRまたは2ステップRT-PCRによって、1本のチューブで最大5つのcDNAまたはgDNA標的を迅速に高い信頼性で定量できます。Q-bondテクノロジーと最適化されたマスターミックスが、1時間以内の超高速マルチプレックスreal-time PCRを促進します。すぐに使える4xマスターミックスに独自のホットスタートとPCRバッファーシステムを組み合わせることで、最適化を必要することなく、あらゆるリアルタイムサイクラーで高感度qPCRが保証され、室温での自動反応セットアップオプションも提供されます。また、正確なピペッティングを視覚的に識別する内蔵トラッキングシステムは、ヒューマンエラーを防ぐのに役立ちます。そして、cDNA合成用のQuantiNova Reverse Transcription Kitと組み合わせることで、QuantiNova Internal Control RNAを組み込んで、逆転写とqPCRの成功をモニターすることができます。
パフォーマンス
QuantiNova SYBR Green PCR Kitを試すのははじめてですか? ぜひトライアルキットの見積りをご依頼ください。
QuantiNova Probe PCR Kitを試すのははじめてですか? ぜひトライアルキットの見積りをご依頼ください。
QuantiNova Multiplex PCR Kitsを試すのははじめてですか? ぜひトライアルキットの見積りをご依頼ください
正確な反応セットアップ用の内蔵視覚的インディケーター
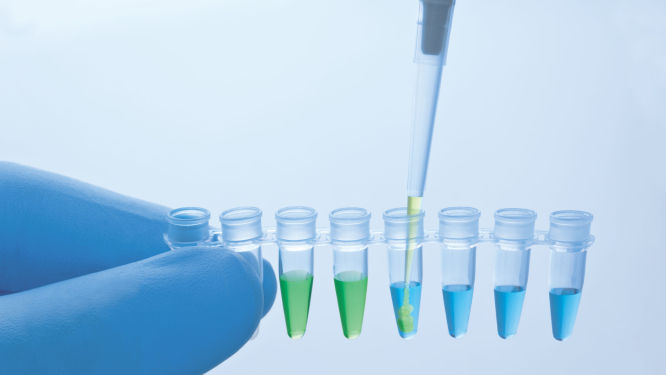
QuantiNova PCR Kitsに付属のマスターミックスには、real-time PCRを阻害しませんが、チューブやウェル内の視認性を高める不活性な青色色素が含まれています。QuantiNova Yellow Template Dilution Bufferには、不活性な黄色色素が含まれています。テンプレート核酸をQuantiNova Yellow Template Dilution Bufferで希釈し、マスターミックスに添加すると、溶液の色が青色から緑色に変化し(図“ 内蔵ピペッティング コントロールによって示される正確な反応セットアップ”参照)、各反応が正しくセットアップされたことを視覚的に示します。
QuantiNova SYBR Green PCR Kit
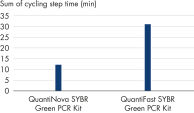
QuantiNova SYBR Green PCR Kitの感度が高いことから、ひとつの標的コピーであっても正確かつ堅牢に検出できます(図“ シングルコピー標的の堅牢な高感度の検出”参照)。この極めて高い感度は、超高速サイクリング条件下でも実現されます。
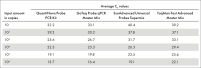
QuantiNova SYBR Green PCR Kitは、幅広いテンプレート量で使用できます。このSYBR real-time PCRキットの性能は、8桁に及ぶダイナミックレンジ(100 ng~10 fg cDNA)でテストされており、使用する機器に関係なく、定量は正確で堅牢でした(図“ 広範囲のダイナミックレンジで正確な定量”参照)。
QuantiNovaケミストリーの厳密な特異性と卓越した感度により、QIAGEN Rotor-Gene Qでは最適な結果が得られますが、フォーマット、高速サイクリング能力、パッシブレファレンス色素の必要性に関係なく、あらゆるreal-time PCRサイクラーで使用できます。必要に応じて、QuantiNova SYBR Green PCR Kitに付属のROXをマスターミックスに加えるだけです。Rotor-Gene Q、Agilent Technologies Mx3005P、Applied Biosystems 7900 HT Fast、ViiA 7、StepOnePlus、7500 Fast、Roche LightCycler 480、Bio-Rad CFX96を使用した増幅と定量の結果は、常に堅牢で高感度です。
QuantiNova SYBR Green PCR Kitは、他のSYBR Green PCRキットに比べ、より優れた結果をもたらします。サイクリング条件や装置に関係なく、QuantiNovaケミストリーは、より低いCT値、より高い再現性、より高い反応効率を生み出すことが実証されています。
QuantiNova Yellow Template Dilution Bufferは、反応セットアップ時に安心感をもたらしますが、QuantiNova SYBR Green PCR Kitで優れた結果を得るために必要というわけではありません。QuantiNovaケミストリーはフレキシブルに設計されているので、確立されたどのようなワークフローにも組み込むことができます。そのため、目的の増幅標的、選択した装置、希釈バッファーを用いて、最適な性能が得られます。
QuantiNova Probe PCR Kit
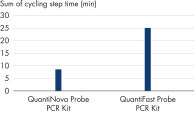
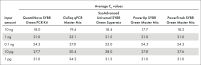
このプローブreal-time PCRキットのreal-time PCRミックスは、30°Cで最大100時間保存可能であり、その後の反応の性能を損なうことはありません。冷却剤を使用せずに室温で長時間保存した後でも優れた安定性を示すため、QuantiNova Probe PCR Kitはハイスループット反応セットアップやプレートスタックの取り扱いに理想的です(表「反応安定性に対する保存の影響」を参照)。
| 平均CT 値 | ||||
| QuantiNova Probe PCR Kit | サプライヤーLのプローブPCRキット | |||
| 量(ng) | 0時間 | 100時間 | 0時間 | 100時間 |
| 10 | 24.37 | 24.49 | 24.35 | 27.49 |
| 1 | 27.78 | 27.90 | 27.77 | 30.97 |
| 0.1 | 31.16 | 30.98 | 31.37 | 34.48 |
| テンプレートなしのコントロール | 不検出 | 不検出 | 不検出 | 不検出 |
30°Cで100時間保存前後のCT値の比較。マスターミックス、テンプレートcDNA、プライマー、TNFのプローブを含むreal-time PCRを30°Cで100時間インキュベートし、新たに調製した反応液とともにreal-time PCRを実行しました。3つの異なるテンプレート量の反応を3連で試験しました。サプライヤーLのプローブPCRキットとは対照的に、QuantiNova Probe PCR Kitが提供するCT値はサンプルを30°Cで長時間保存しても変化しませんでした。
QuantiNova Probe PCR Kitの感度が高いことから、ひとつの標的コピーであっても正確かつ堅牢に検出できます(図“ ひとつの標的コピーの高感度かつ堅牢な検出”参照)。これらキットに付属する特別なマスターミックスが、1本のチューブで、存在量が大きく異なる2つの標的の正確な定量を可能にします。これにより、時間と費用を節約し、必要なサンプル量を減らすことができます。さらに、得られるデュプレックスPCRデータはシングルプレックスPCRで得られるデータと同等です。
QIAGEN独自の実績のあるバッファー技術と新しいQuantiNova DNA Polymerase、QuantiNova Antibody、QuantiNova Guardの独自の組み合わせにより、困難なreal-time PCRアッセイであっても、コストと時間のかかる最適化を行うことなく、初回の試行でreal-time PCRを確実に成功させることができます(図“ 困難なアッセイでの優れた結果”参照)。
QuantiNova Probe PCR Kitはどのようなリアルタイムサイクラーでも使用できます。ROXは別のチューブで提供され、ROXをパッシブレファレンス色素として使用するサイクラーを使用する場合は、追加することができます。このキットは、感度、再現性、効率に関して、さまざまなサイクラー間で非常に一貫性のある結果を提供します。さまざまなサイクラーの高速サイクリング能力が異なるため、サイクリングプロトコールが異なるにもかかわらず、結果の一貫性は維持されます。
QuantiNova Multiplex PCR Kits
QuantiNova Multiplex PCR Kitsに付属する特別なマスターミックスは、マルチプレックス反応の迅速なセットアップを可能にし、初回の試行で成功した結果をもたらし、シングルプレックスPCRデータと同等のマルチプレックスPCRデータを提供します。高濃度の4xマスターミックスは最大800 ngのテンプレートインプットに対応し、最大5プレックス反応でも卓越した感度を保証します。このキットはテンプレート量のわずかな違いを明確に区別することができ、存在量が大きく異なる標的を正確に定量します。
抗体を介した新しいホットスタートメカニズムにより、卓越した特異性が保証され、室温での反応セットアップが可能になるため、自動化された手順に理想的です。特別に開発された高速PCRバッファーには添加剤Q-Bondが含まれており、これは、アニーリングと伸長時間を大幅に短縮し、マルチプレックスqPCRを1時間未満で行うことができます。視覚的ピペッティングコントロールは、特に定量的2ステップRT-PCR用のQuantiNova Reverse Transcription Kitに付属のInternal Control RNAと組み合わせると、ヒューマンエラーを防ぎ、プロセスの安全性を高めるのに役立ちます。
QuantiNova Multiplex PCR Kitは、超高速かつインプロセスで制御されたマルチプレックスqPCRを使用して、限られたサンプル材料からより多くの洞察を得ることで、ワークフローの効率を向上させます。
図参照
原理
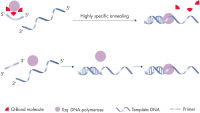
QuantiNova PCR Kitsは、新しい抗体を使ったホットスタートメカニズムにより、cDNAやgDNA分析を最高の特異性で実現します(図“ 新しいQuantiNovaホットスタートメカニズムの原理”参照)。低温では、QuantiNova DNA Polymeraseは、QuantiNova Antibodyおよび複合体を安定化させる新しい添加剤QuantiNova Guardによって不活性状態を維持します。これにより、ホットスタートの厳密性が改善され、非特異的にアニーリングされたプライマーとプライマーダイマーの伸長を防ぎます。温度を95°Cに上げると、2分以内にQuantiNova AntibodyとQuantiNova Guardは変性し、QuantiNova DNA Polymeraseが活性化されて増幅が可能になります。
さらに、QuantiNovaケミストリーは、PCRの感度と効率を損なうことなくワークフローを合理化する機能を備えています。マスターミックスとQuantiNova Yellow Dilution Bufferに含まれる不活性色素は、各反応が正しくセットアップされていることを視覚的に示すインジケーターとして機能します。マスターミックスの青色は反応容器内の内容物の視認性を高め、QuantiNova Yellow Dilution Bufferで希釈したテンプレート核酸を添加すると緑色に変わります(図“ 内蔵ピペッティングコントロールによって示される正確な反応セットアップ”参照)。QuantiNova SYBR PCR Kitsに含まれるPCRバッファーの独自の配合により、ホットスタートメカニズムの厳密性が強化され、サイクリングステップが短縮されるため、PCRがより迅速に実行され、スループットが向上します。
QuantiNova Multiplex PCR Kitsは、標準サイクラーと高速サイクラーの両方で、最適化しなくても、広いダイナミックレンジで高感度かつ迅速な結果を提供します。特別に開発された高速PCRバッファーには、アニーリングと伸長時間を大幅に短縮する添加剤Q-Bondが含まれています(図「 高速プライマーアニーリング」参照)。
レファレンス遺伝子と標的遺伝子を別々の反応ではなく、同じ反応で増幅すると、取り扱いエラーが最小限に抑えられ、遺伝子定量の信頼性が向上します。さらに、定量的2ステップRT-PCR用のQuantiNova Reverse Transcription Kitに含まれるInternal Control RNAを組み込むことで、逆転写とqPCRの成功をモニターできます。
QuantiNova Multiplex PCR Bufferは、K+イオンとNH4+イオンがバランスよく組み合わされているだけでなく、独自の合成Factor MPが含まれており、これらがともに、プライマーとプローブの核酸テンプレートへの安定的かつ効率的なアニーリングを促進し、高いPCR効率を可能にします(図「 独自のマルチプレックスPCRバッファーが安定的かつ効率的なアニーリングを促進」参照)。
QuantiNova Multiplex PCR Kitのマスターミックスは2–8°Cで最大12ヶ月間便利に保存でき、また反応セットアップは室温で極めて安定しているため、効率と精度を高める自動化手順が可能になります。
図参照
操作手順
QuantiNova PCR Kitsにはすぐに使用できるマスターミックスが含まれており、反応条件やサイクリング条件を最適化する必要がありません。マスターミックスにテンプレートgDNAまたはcDNAとプライマー(SYBR Greenベースの検出)またはテンプレートgDNAまたはcDNAとプライマーとプローブ(プローブベースの検出)を加え、ハンドブックのプロトコールに従うだけで、あらゆるリアルタイムサイクラーで迅速に信頼性の高い結果が得られます。ROXパッシブレファレンス色素は別のチューブで提供され、使用するサイクラーのタイプに応じて色素濃度を調整できます。したがって、QuantiNova PCR Kitsは、形式やサイクリング条件に関係なく、事実上どのようなリアルタイムサイクラーでも使用できます。最適化されたROX濃度により、低コピー数であっても自動データ分析によって検出が可能です。
QuantiNova Multiplex PCR Kitsにはすぐに使える4xマスターミックスが含まれており、反応条件やサイクリング条件を最適化する必要がありません。最大800 ngのテンプレートDNAとプライマープローブセットをマスターミックスに加え、ハンドブックのプロトコールに従うだけで、あらゆるリアルタイムサイクラーで迅速に信頼性の高い結果が得られます。キットにはROXパッシブレファレンス色素が別のチューブで提供され、使用する装置に応じて、適切なROX濃度に調整できます。
リアルタイム2ステップRT-PCRで最適な結果を得るには、ゲノムDNAコンタミネーションの除去が組み込まれ、わずか20分でcDNAを高速合成するQuantiNova Reverse Transcription Kitを用いてcDNAを合成することをお勧めします。さらに、逆転写とqPCRの成功をインプロセスでモニターすることができるQuantiNova Internal Control RNAも提供します。
効率的なワークフローにするには、QuantiNova Kitsを当社の設計済みのQuantiNova real-time PCRアッセイやパネルと組み合わせることをお勧めします。これは、存在量に関係なく、mRNAや長鎖ノンコーディングRNA転写産物を正確に定量します。幅広い設計済みのヒト、マウス、ラットのプライマーセットから選択するか、当社の高度な設計ツールを使用してご自身のアッセイやパネルをカスタマイズすることができます。
当社のQuantiNova LNA PCRおよびQuantiNova LNA Probe PCR Assaysは、LNAテクノロジーを利用して感度を向上させ、偏りのない遺伝子発現プロファイルと迅速な科学的洞察を可能にします。
アプリケーション
QuantiNova SYBR Green PCR Kitは、あらゆるリアルタイムサイクラーで、SYBR GreenベースのcDNA標的の遺伝子発現分析や定量的gDNA分析に使用できます。これには、Applied Biosystems、Bio-Rad、Cepheid、Eppendorf、LIFE Technologies、Roche、Agilentの装置やRotor-Gene Qが含まれます。
QuantiNova Probe PCR Kitsは、あらゆるリアルタイムサイクラーで、cDNA標的のプローブベースの遺伝子発現分析や定量的なgDNA分析に使用できます。これにはApplied Biosystems、Bio-Rad、Cepheid、Eppendorf、Roche、Agilentの装置が含まれます。
QuantiNova Multiplex PCR Kitsは、あらゆるリアルタイムサイクラーで、cDNAまたはgDNA標的のマルチプレックス遺伝子発現分析に使用できます。マルチプレックスの利点を最大限に活用するには、Rotor-Gene Qのような最大5プレックスキャパシティを提供する装置をお勧めします。
裏付けデータと数値
ピペッティングコントロールによって示される正確な反応設定。