低頻度変異を確実に検出可能なデジタルターゲット
QIAseq Targeted DNA Panel (12)
Cat. No. / ID: 333502
特徴
- 分子バーコードによるPCR duplicate や偽陽性排除が可能
- 迅速かつシンプルなワークフローでライブラリー調製を実現
- ヒト由来のFFPE や遊離DNA にも対応可能
- 10 ng からの微量サンプルに対応
- 高GC 領域でのカバレッジを最大限にするためのプライマーデザインとbuffer テクノロジー
製品詳細
QIAseq targeted DNA Panels は、分子バーコードによるデジタルシークエンスを可能にするターゲットシークエンス用ライブラリー調製試薬です。分子バーコードによりPCR duplicate、偽陽性およびライブラリー増幅バイアスの問題を克服し、正確な低頻度変異検出を可能にします。また、GeneGlobe によるクラウドベースのフリーデータ解析ツールを提供しています。本製品はターゲット増幅からライブラリー構築に必要な試薬が1キットに全て含まれています。Single primer extension(SPE)技術による特殊なプライマーデザインで、1種類の遺伝子特異的なプライマーでターゲット増幅が可能なため、必要なプライマー数は従来のPCR 増幅法の半分で済み、1チューブでご提供が可能です。さらに、SPE 技術はデザインのフレキシビリティを格段に向上させ、難しい領域へのプライマーデザインも可能にします。サンプルインデックスは、ご使用のプラットフォームに合わせて適切なキットを別途ご選択いただき、組合せで最大384マルチプレックスまで可能です(イルミナおよびサーモフィッシャーシークエンサー対応)。
パフォーマンス
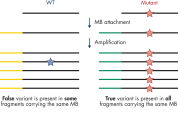
- 精確さ:革新的なデジタルシークエンシング(分子バーコードを含む)により、PCR duplicateと増幅アーティファクトが排除され、低頻度な変異を高い信頼性をもって検出できます(図:分子バーコードの原理 参照)。
- 特異性:独自のアルゴリズムによるプライマーデザインによる高い特異性と正確な結果をもたらします。
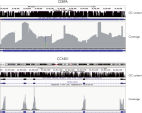
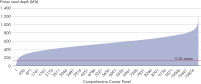
- Uniformity:QIAseq Targeted DNA Panelsのワークフローは、シークエンシングキャパシティーを最大限に利用し、カバレッジ均一性の高いシークエンシング結果が得られるよう最適化されています(図:Uniformity 参照)。
- 感度:デジタルDNA シークエンシング法は、低頻度なDNA 変異を高い信頼性で同定できるように最適化されています。各遺伝子のコード領域全体をカバーするプライマーデザインの場合、アリル頻度1% の NA12878 SNPと典型的なコード領域のインデルに対して90%以上の感度で検出可能で、偽陽性の検出率は15塩基/100万塩基以下となっています。
- 普遍性:QIAseq Targeted DNA Panelsは、ユニークなプライマーデザインアルゴリズム(SPE法)により、通常のゲノム領域とGCリッチで解析が困難なゲノム領域両方に対応しており、CEBPAやCCND1などのGC含有率の高い遺伝子を100%カバーします(図:GCリッチなゲノム領域のカバレッジ 参照)。
- フレキシビリティ:QIAseq Targeted DNA Panels は、柔軟性に富んだコンテンツで、デザイン済みのカタログパネルは、様々ながん腫にご使用いただけます。また、新規にカスタムパネルを作製することも、既存のカタログパネルに、100プライマーまでプライマーを追加して内容を拡大することもできます。さらに、フレキシブルなサンプルマルチプレックスが可能で、最大384サンプルまで、QIAseq インデックスを用いてマルチプレックスが可能です。
原理
PCR duplicate はターゲットシークエンシングの大きな問題の一つです。その理由は、PCR 増幅を通じて、固有であったDNA 分子を互いに区別できない同じDNA 分子に変えるからです。さらに、PCR 増幅とシークエンシングプロセスで生じるエラーは最終リードにも存在し得るため、偽陽性と真の変異の識別ができず、DNA に低頻度で存在する変異を確実に検出できなくなる可能性があります。PCR duplicate と増幅アーティファクトの問題を解決するため、QIAseq Targeted DNA Panels は、分子バーコードを増幅前のDNA に組み込んでから増幅を行います。これによりスタート時点でのDNA 分子の固有性を維持することが可能になり、PCR duplicate、偽陽性、ライブラリーバイアスの問題を克服します。
操作手順
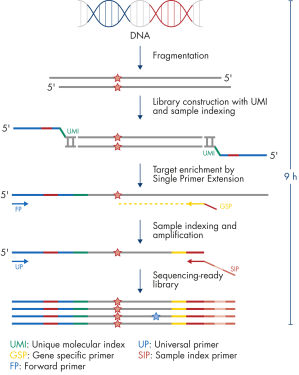
QIAseq Targeted DNA Panels の全ワークフローは約9時間で、抽出したDNAからシークエンシング可能なライブラリー調製まで完了します(図:ワークフロー 参照)。抽出したDNAを断片化後、分子バーコードを付加し、ターゲットを濃縮し、ライブラリーを調製します。シークエンシングファイルは、キアゲンのクラウドベースのデータ解析パイプライン、GeneGlobe に組み込むことができます。これは、リードをフィルタリング、マッピング、アラインメントするステップを含むだけでなく、ターゲット領域の分子バーコードをカウントし、分子バーコードに基づいた変異を識別することもできます。出力されたデータは、弊社のデータ解釈ツールQCI にインポートすることも可能です。
アプリケーション
QIAseq Targeted DNA Panels は、多様なアプリケーションとサンプル種に対応しており、様々なDNA 変異を検出できます。
DNA variants:
- SNVs
- Small indels
- CNVs
サンプルの種類:
- FFPE
- 血漿/血清
- 新鮮な組織または凍結組織
- 細胞
アプリケーション:
- 固形腫瘍や血液腫瘍のDNA変異のプロファイリング
- 固形腫瘍におけるホットスポットの検出
- ミトコンドリアDNAの変異検出
- 疼痛およびADMEファーマコゲノミクス
- ヒト個人識別と父子鑑別
- 遺伝性疾患についての生殖細胞系列突然変異の評価
- BRCA1 とBRCA2 の全エクソンのプロファイリング
裏付けデータと数値
ワークフロー
最小10 ng の精製DNAを酵素法により断片後、IL-N7アダプター、分子バーコード、片側のサンプルインデックスを付加します。つづいてSingle Primer Extension(SPE)によるターゲット濃縮を行います。SPE 法は、アダプター配列をユニバーサルフォワードプライマーとして利用することにより、遺伝子特異的プライマーは片側のみでターゲット濃縮できる技術です。最終的に、IL-S5サンプルインデックスプライマーとユニバーサルプライマーを用いてライブラリー増幅を行います。その際、同時にもう一方のサンプルインデックス付加(dual index 対応)が行われ、ライブラリー構築が完成します。