REPLI-g Single Cell Kit
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
REPLI-g Single Cell Kit (24)
Cat. No. / ID: 150343
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特徴
- シングルセルからの最大のゲノムカバレッジでの全ゲノム増幅
- MDA テクノロジーによりゲノム遺伝子座の偏りのない増幅
- 次世代シークエンシングのような新テクノロジーでの使用に最適
- 最大40 μg までの一定した収量(増幅産物長さの平均は>10 kb)
- がん研究やメタゲノミクスなど、多くのアプリケーションに最適なツール
製品詳細
次世代シークエンシング(NGS)などの革新的テクノロジーを用いてDNA配列解析やジェノタイピングを行なう場合などは限られたサンプル量しか用いることしかできない場合が数多くあります。REPLI-g Single Cell Kitはシングルセル(1,000 個未満の細胞から最低1 個の細菌または腫瘍細胞まで)あるいは精製ゲノムDNAから配列の偏りがなく、最大のゲノムカバレッジで均一にゲノムDNAを増幅するようにデザインされています。新鮮または凍結された組織、および新鮮または乾燥した血液を用いるための追加プロトコールもあります。専用のバッファーと試薬は、ユニークで制御された除染プロセスは、混入DNAの増幅を防ぎ、毎回の信頼性の高い結果を保証します。ごくわずかな配列の偏りとゲノムの欠落がない正確なゲノム増幅が画期的なMultiple Displacement Amplification (MDA)を用いて実現します。 PCR ベースのWGA テクノロジーとくらべて最高1,000 倍の高いフィデリティを示し、偽陽性や陰性の結果を排除します。WGA テクノロジーについては、弊社ウェブサイトの全ゲノム増幅に関する情報ページをご覧になるか、REPLI-g 製品については表4をご参照ください。
パフォーマンス
完全なゲノムカバレッジでNGSやその他のダウンストリームアプリケーションに最適
REPLI-g Single Cell Kitを用いて増幅したDNAの長さは平均 >10 kbであり、カバレッジを最大限にしました。 本キットは次世代シークエンシング(NGS)、array comparative genomic hybridization(aCGH)、リアルタイムPCRを用いたアプリケーションを含む様々なダウンストリーム解析でテストされており、これらの用途に最適なキットです(表2)。PCRを利用した増幅ステップを別途に行なう必要がないため、REPLI-g全ゲノム増幅とライブラリ調製にかかる手作業時間を短縮でき、その結果、PCRベースの方法よりも長いリードが得られます (図 " REPLI-g増幅DNAを用いた次世代シークエンシングは、PCRベースの方法よりも手作業時間が短く多くの配列情報を生成")。精製したゲノムDNAまたはREPLI-g Single Cellで増幅したDNAの両方で、わずか1個のバクテリア細胞を用いた場合でも、同等の高品質なNGS結果が得られ、高い配列カバー率および非常に低いエラー率を示しました(図 “ NGSで同等の結果を実現”)。これらの結果は、すべてのヒト常染色体とX染色体をカバーする広範囲のマーカーを用いた網羅的な解析によっても示され、3つの異なる独立した実験は、ゲノムの全領域からDNAが良好に増幅されたことを実証しています(図 “ 高いゲノムカバレッジ”)。
| サンプル(細胞/DNA) | 研究分野 |
|---|---|
| ヒト/動物 | バイオマーカー研究(SNPs、変異、CNV) |
| 幹細胞研究 | |
| 胎児由来の循環細胞解析 | |
| モザイク遺伝研究 | |
| 遺伝的素因の研究 | |
| トランスジェニック動物のタイピング | |
| がん | 体細胞変異解析 |
| 腫瘍進行 | |
| 腫瘍幹細胞/進化 | |
| 循環腫瘍細胞の解析 | |
| バクテリア | メタゲノム研究 |
| 病原体解析 | |
| 微生物ジェノタイピング | |
| 植物* | 気孔研究 |
| 花粉解析 |
他社に比べて卓越した性能のREPLI-g Single Cell Kit
他のメーカーが通常採用しているPCRベースのWGA法は、ダウンストリームのプロセスに影響する可能性があるPCRプライマー配列を末端にもつ短い断片を生成します(例;次世代シークエンシング;図" REPLI-g増幅したDNAを用いた次世代シークエンシングは、PCRベースの方法よりも手作業時間が短く多くの配列情報を生成")。PCRベースのWGA法は、エラーを発生しやすい増幅(結果として単一塩基対変異、STRの短縮および伸長)、またフィデリティが低い酵素Taq DNAポリメラーゼによる遺伝子座の偏りや低い再現性につながることがあります。これとは対照的に、REPLI-g Single Cell Kitはゲノム全体にわたり均一性の高い増幅を実現し、増幅中の遺伝子座の偏りは最小限に抑えられます。REPLI-g Single Cell Kitを含む2種類のMDAテクノロジーを利用したキットおよび2種類のPCRベースのキットの合計4種類のWGAキットで、各キットに特化されたシングルセル増幅プロトコールを用いて配列再現性と遺伝子座の脱落をテストしました。REPLI-g Single Cell Kitとは異なり、他社のキットを用いて解析したシングルセルでは、完璧で偏りのない配列再現性はほとんどの場合得られませんでした(図 “ シングルセルから信頼性が高く偏りのないDNA 増幅を実現”)。
図参照
原理
REPLI-g Single Cell Kitに含まれるREPLI-g sc Polymeraseは、Multiple Displacement Amplification (MDA)テクノロジーを用いて複雑なゲノムDNAを増幅するために、革新的なハイフィデリティ酵素Phi29 ポリメラーゼを最適化済みで、DNAの断片化や脱塩基部位の形成を防ぐために緩和なアルカリ変性ステップと組み合わせて使用します。本キットは分離したがん細胞やバクテリアなどのシングルセルから高収量でDNAを増幅するために特化されています(表1)。さらに、様々な臨床および非臨床研究サンプルや精製ゲノムDNAと共に使用することができる一方、鮮血、乾燥血液や新鮮・凍結組織で使用する追加プロトコールも入手可能です。典型的なDNA収量は、スタートテンプレート量に関係なく一貫して40μgに達し、DNA濃度を測定せずにその後の遺伝子解析を実施できます。増幅産物の平均長が10 kbを超え完全なゲノムカバレッジを持つことから、REPLI-g Single Cell Kitで増幅したDNAは、次世代シークエンシング(NGS)、アレイを利用したComparative Genome Hybridization(アレイCGH)、パイロシークエンス法、リアルタイムPCR解析(表2)などの様々なアプリケーションに最適です。
| アプリケーション | 装置 |
|---|---|
| 全ゲノムシークエンス | 次世代シークエンスプラットフォーム† |
| エクソームシークエンス | |
| SNPジェノタイピングアレイ | アレイプラットフォーム† |
| アレイCGH | |
| qPCR/PCR テクノロジー | リアルタイムPCR/PCR サイクラー† |
| サンガーシークエンス | キャピラリーシークエンサー† |
| パイロシークエンス | PyroMark(QIAGEN) |
増幅原理
REPLI-g Single Cell KitはMultiple Displacement Amplification(MDA)と呼ばれる等温ゲノム増幅を使用しています。これは変性DNAにランダムヘキサマーを結合し、次に最適化された強い鎖置換の特性を持つPhi29 ポリメラーゼを用いて一定の温度で鎖置換合成を行ないます。テンプレートとして機能する各置換鎖で追加のプライミングが起こり、高収量の増幅DNAが得られます(図“ Multiple Displacement Amplification(MDA)technology”)。ファージ由来の酵素であるPhi 29 ポリメラーゼは、3 '→5'エキソヌクレアーゼ活性(プルーフリーディング活性)を有するDNAポリメラーゼで、Taq DNAポリメラーゼに比べて1000倍も高いフィデリティを実現します。ユニークで最適化されたREPLI-g Single Cellバッファーにより、Phi29ポリメラーゼはヘアピンループなどの二次構造を容易に解離し、それによって増幅中のポリメラーゼのスリッピング、停止、解離を防止します。これにより100 kbまでのバイアスのないDNAフラグメントの生成が可能です(図" Phi29ポリメラーゼでバイアスのない増幅”)。
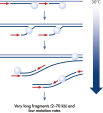
細胞溶解とDNAのアルカリ変性
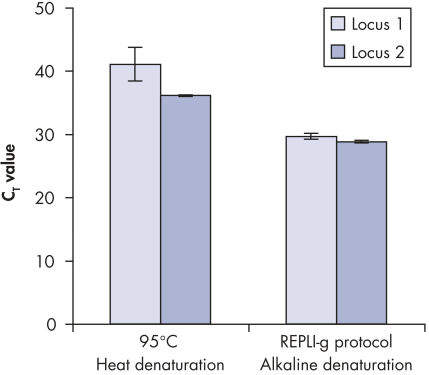
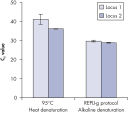
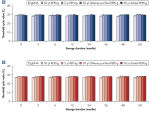
酵素による増幅を行なう前にゲノムDNAは高温(熱インキュベーション)または高pH(化学的アルカリ性インキュベーション)でのインキュベーションのような過酷な方法を使用して変性させる必要があります。REPLI-g Single Cell Kitは穏やかなアルカリ性インキュベーションを使用し、細胞の溶解とDNAのフラグメント化や脱塩基部位が非常に少ない均一なDNA変性を可能にします。この結果、非常に高い整合性をもつ増幅DNAが得られ、増幅されたフラグメントの長さを最大限に引き出し、ゲノム遺伝子座と配列が均一に表現されます(図" 熱およびアルカリによる変性の遺伝子座への影響")。
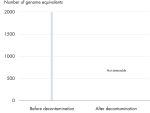
検出可能なDNAコンタミを効率的に除去
すべてのREPLI-g Single Cell Kit は、REPLI‑g で増幅可能なDNAコンタミ除去を確実にするため制御された独自の方法で夾雑核酸を取り除きます。バッファーおよび試薬は、検出可能な残留コンタミDNAが存在しないことを確保するため、革新的で標準化された方法によりUV照射を実施しています(図 “ 画期的な残留DNAの除去”)。このUV処理後に、完全な機能性を確実にするためにキットの品質管理を厳密に行ないます。
図参照
操作手順
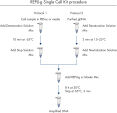
簡便な1チューブでの操作
REPLI-g Single Cell Kitは簡便で信頼できるシングルセルや限られた量のサンプルからの正確なゲノム増幅を行なえます。 簡便な反応セットアップとわずか15分のマニュアルでの作業時間により、わかりやすく信頼できる手法となっています(図“ REPLI-g Single Cell Kit操作手順”)。 専用のバッファーおよび試薬は、シングルセル、限られた量の組織サンプル、精製DNAから、高い配列再現性と偏りのない増幅で高純度な全ゲノム増幅DNAを実現するように開発されています(表3)。 REPLI-gで増幅されたDNAは–20°Cで問題なく長期保存できます(図 " Consistent long-term stability")。 REPLI-g製品のラインナップと特徴の概略は表4をご参照ください。
| キット構成品 | 利点 |
|---|---|
| REPLI-g sc Polymerase | 最長70 kbまでの長いフラグメント |
| Taqよりも1,000倍高いフィデリティ | |
| 高い配列再現性 | |
| 全遺伝子座で均一な増幅 | |
| REPLI-g sc Reaction Buffer‡ | 偏りのない増幅と全遺伝子座の再現に最適化 |
| Buffer DLB(溶解と変性) | 増幅のための効率的な調製 |
| DNAを損傷しないプロセス | |
| UVによるコンタミ除去プロセス | 検出可能な残留DNAコンタミを確実に除去 |
| REPLI-g Single Cell | REPLI-g Mini | REPLI-g UltraFast Mini | REPLI-g Midi | REPLI-g Screening | REPLI-g FFPE | REPLI-g Mitochondrial DNA | |
|---|---|---|---|---|---|---|---|
| スタートサンプル | シングルセル、gDNA | 精製gDNA、血液、細胞 | 精製gDNA、血液、細胞 | FFPE組織、FFPE組織から精製したgDNA | 精製gDNA | ||
| (その他のスタートサンプルのプロトコールはwww qiagen.comをご覧ください) | |||||||
| 使用量 | シングルセル、2~1000個、組織、精製gDNA(1~10 ng) | >10 ng gDNA、0.5 µl 血液または細胞(>600 細胞/µl) | >10 ng gDNA、0.5 µl 血液または細胞(>600 細胞/µl) | 切片(1 cm diamter, 10~40 µmの厚さ); >100 ng gDNA | >1 ng 精製gDNA | ||
| 収量(µg/reaction) | 40 | 10 | 7~10 | 40 | 8 | 標準収量:≤10;高収量:≤40 | 3~5 |
| 反応時間(時間) | 8~16 | 10~16 | 1.5 | 8~16 | 12~16 | 標準収量:4 ; 高収量:10 | 8 時間 |
| 作業時間(分) | 15 | 15 | 15 | 15 | 15 | 40 | 15 |
| フォーマット | チューブ | チューブ | チューブ | チューブ | プレート | チューブ | チューブ |
図参照
アプリケーション
REPLI-g Single Cell Kitを用いて増幅したDNAは以下のダウンストリームアプリケーションに使用できます:
- 次世代シークエンシング
- TaqManプライマー/プローブセットを用いたSNPジェノタイピング
- qPCR および PCRベースの変異解析
- STR/マイクロサテライト解析
- サンガーシークエンス
- パイロシークエンス
- Comparative Genome Hybridization(CGH法)などのアレイテクノロジー
裏付けデータと数値
Effect of heat and alkaline denaturation on loci representation.
Effect of heat and alkaline denaturation on loci representation.