✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
MinElute Reaction Cleanup Kit (50)
Cat. No. / ID: 28204
✓ オンライン注文による24時間年中無休の自動処理システム
✓ 知識豊富で専門的な製品&テクニカルサポート
✓ 迅速で信頼性の高い(再)注文
特徴
- 非常に少量の溶出液量
- 迅速かつ簡単な操作
- 再現性と高い回収率
- サンプル操作を便利にするローディングダイ
製品詳細
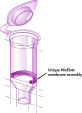
MinElute Reaction Cleanup Kitは、酵素反応液から70 bp – 4 kbサイズのDNAを精製するためにシリカメンブレンスピンカラム、バッファー、およびコレクションチューブを提供します。スピンカラムは、僅かな量(10 µl)で溶出、高濃縮されたDNAを高い収率で得ることができます。組み込まれたpH指示薬により、スピンカラムへのDNA結合の至適pHを容易に確認できます。この操作は、QIAcube Connectで完全自動化できます。MinEluteシステムで精製したDNAフラグメントは、シークエンシング、マイクロアレイ解析、ライゲーションと形質転換、制限酵素消化、標識、マイクロインジェクション、PCR、およびin vitro転写を含むすべてのアプリケーションで使用できます。
最適な結果を得るには、本製品をQIAvac 24 Plusと併用することをお勧めします。
パフォーマンス
MinElute Reaction Cleanup Kitは、酵素反応液から最大5 µgのDNA(70 bp ~ 4 kb)のクリーンアップを保証し、広範なアプリケーションに適した高収量のDNAを得ることができます。このキットは、酵素反応液のクリーンアップのためのスピンカラムを提供します。小型遠心機または真空マニホールドを使用して、高濃度のDNAフラグメント(70 bp – 4 kb)を迅速に得ることができます。(4 kbより大きいDNAフラグメントは、QIAquick Systemを使用して精製してください。)
| タンパク質 | Molecular weight per enzyme subunit (kDa) |
|---|---|
| DNA Polymerase I | 109 |
| Klenow fragment | 62 |
| 子牛小腸アルカリンホスファターゼ | 69 |
| T4 DNA ligase | 55 |
| T4 Polynucleotide kinase | 35 |
| Terminal transferase | 32 |
| DNase I | 31 |
| 制限酵素 | 多様 |
原理
MinElute Kitsは、高塩濃度バッファーでDNAを結合し、低塩濃度バッファーまたは水で溶出するためのシリカメンブレン技術です。この精製操作は、DNAサンプルからプライマー、ヌクレオチド、酵素、ミネラルオイル、塩、アガロース、臭化エチジウムなどの不純物を除去します。シリカメンブレン技術では、緩い樹脂やスラリーに伴う問題や不都合がありません。特殊な結合バッファーは、特定のアプリケーション向けに最適化され、特定のサイズ範囲内でのDNA分子の選択的吸着を促進します。
Gel loading dye
ローディングダイは、迅速で便利なサンプル処理と分析を可能にします。GelPilot Loading Dyeは、3種類のトラッキングダイ(キシレンシアノール、ブロモフェノールブルー、およびオレンジG)を含み、アガロールゲルのランタイムの最適化を容易にし、より小さなDNAフラグメントの過度な移動を防ぎます(「 GelPilot Loading Dye」の図を参照)。
図参照
操作手順
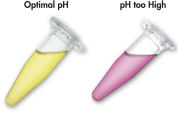
MinEluteシステムは、シンプルな結合-洗浄-溶出の手順です(「 MinEluteの操作手順」のフローチャートを参照)。結合バッファーを、酵素反応液に添加し、混合液をMinEluteスピンカラムにアプライします。結合バッファーはpH指示薬を含んでおり、DNA結合の至適pHを容易に確認できます( 「pH Indicator Dye」の図を参照)。核酸は、バッファー由来の高塩濃度条件下でシリカメンブレンに吸着します。不純物は洗い流され、純粋なDNAが、少量の低塩濃度バッファーまたは水と共に溶出し、後のアプリケーションに使用できます。
取り扱い
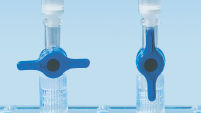
MinEluteスピンカラムは、2通りの便利な操作が可能です(「MinEluteの操作手順」のフローチャートを参照)。スピンカラムは、小型遠心機またはQIAvac Luer Adapters付きのQIAvac 24 PlusやQIAvac 6Sなどのルアーコネクター付きの真空マニホールドにフィットします。 MinElute Reaction Cleanup Kitは、その他のQIAGENスピンカラムキットに加えて、QIAcube Connectで 完全自動化し、生産性の向上、結果の標準化することができます(「スピンカラムの取り扱いオプション A、 B、 C、 D、 E」および「 QIAcube Connect」の図を参照)。
図参照
アプリケーション
MinEluteシステムで精製したDNAフラグメントは、以下を含むすべてのアプリケーションで使用できます。
- シークエンシング
- マイクロアレイ解析
- ライゲーションと形質転換
- 制限酵素処理
- Labeling
裏付けデータと数値
スピンカラム取り扱いオプション — E。

Specifications
| Features | Specifications |
|---|---|
| Binding capacity | 5 µg |
| Fragment size | 70 bp ~ 4 kb |
| Processing | Manual |
| Elution volume | 10 µl |
| Recovery: oligonucleotides dsDNA | 回収:オリゴヌクレオチド、dsDNA |
| Removal <10mers 17–40mers dye terminator proteins | 除去<40mers |
| Format | チューブ |
| Sample type: applications | DNA、オリゴヌクレオチド:酵素反応 |
| Technology | シリカテクノロジー |