Products
特徴
- 130万以上のアッセイが、Ensemblのヒト、マウス、ラットのmRNAおよびlncRNA転写産物を最も幅広くカバー
- LNAプライマーとFAMラベルの加水分解プローブによる卓越した感度と特異性
- 1 RNAコピーから始まる広いダイナミックレンジにわたる正確な検出
- 室温での安定性がもたらす自動化対応への柔軟性
- QuantiNova Probe PCR Kit により、わずか2時間で再現性のある結果
製品詳細
QuantiNova LNA PCRアッセイは、加水分解プローブベースのqPCR遺伝子発現分析による新しいスタンダードです。 一般的な転写産物の検出から特定の転写産物のアイソフォームまで、ヒト、マウスおよびラットのmRNAまたはlncRNAを正確かつ高感度に検出できるように、あらかじめデザインされた幅広いアッセイをご用意しました。 LNA を組み込むことで、プライマーをより短くできるため、柔軟にターゲット上に設計ができるようになりました。その結果、特定の転写産物の検出や近似したターゲットを区別して定量することができます。 さらに、徹底したデザインの検証により、最適なパフォーマンスと優れた検出を保証します。またメジャーなmRNAやlncRNAのアッセイは、ウェットラボにて検証済みです。QuantiNova試薬で最適化されたシンプルなQuantiNova LNA PCR ワークフローは、わずか2時間で結果を手に入れられます。また室温でのセットアップが可能なため、自動化にも対応し、装置の運転終了を待つ作業計画の余裕も提供します。
製品の詳細については、弊社技術スタッフにお気軽にお問い合わせ ください。
パフォーマンス
LNA によるqPCR パフォーマンスの向上
QuantiNova LNA プローブPCRアッセイは、厳しい設計基準とラボで検証されたアルゴリズムを適用しています。 各厳密なアッセイ選択プロセスにより、信頼と正確な結果をゴールとした、特異的かつ増幅効率のよいプライマー・プローブセットが完成します。Tm の標準化とLNA による強化 により、プライマーは標準的なDNA プライマーよりも高い結合親和性が得られ、アッセイの感度と特異性が劇的に向上します。
検出感度を向上させたQuantiNova LNA プローブPCR アッセイは、1 RNA分子までの優れた増幅効率が保証され、微量のサンプルからlncRNAなどの少量のターゲットをより簡単に検出できるようになります(図 QuantiNova LNA Probe PCR Assays provide accurate, sensitive and linear quantification of targets over a wide dynamic range と QuantiNova LNA Probe PCR Assays enable both high-expression and low-expression detection of mRNA 参照)。LNAの絶妙な配置で特異性の向上と高いS / N比を獲得し、1塩基のみ異なる配列の識別と、非特異的な増幅とプライマーダイマーの形成が排除されます。
LNA 塩基の高い結合親和性により、転写物上でのプライマーとプローブの配置の柔軟性が高まるため、設計の自由度が向上し、他の方法では難しいターゲットのアッセイを構築できます。これにより、AUが豊富なターゲット、存在量の少ない転写産物、高い二次構造を持つターゲット、非常に複雑なサンプルであっても、ターゲットの識別と信頼性の高い定量が可能になります。
QuantiNova 試薬とのベストマッチ
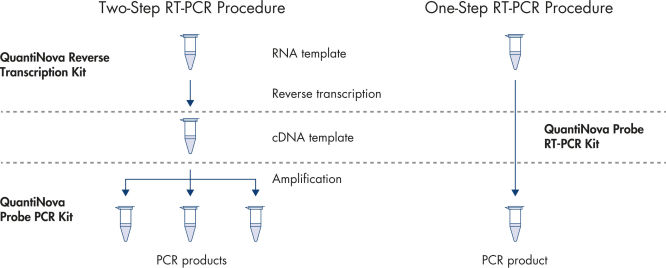
QuantiNova LNA プローブPCR アッセイは、QuantiNova PCR 試薬と共に使用した場合に最適な結果と再現性を提供するように開発されました(図 Demonstrated lot-to-lot reproducibility 参照)。1ステップqRT-PCRの場合はQuantiNova Probe RT-PCRキットが、また2ステップqRT-PCRにはQuantiNova Reverse TranscriptionキットとQuantiNova Probe PCRキットが最適です。
QuantiNovaのホットスタート機構により、GC含有量が高いテンプレートや複雑な二次構造などの難しいテンプレートでも、安心してqPCRを実施できます。また、lncRNA 解析にも最適です(図 Consistently high efficiencies throughout the expected GC content range of the human transcriptome 参照)。ホットスタートは、室温でのサンプル調製を可能にし、ロボットによる分注の自動化やサンプル調整後も余裕をもって装置にセットすることができます。さらに、視覚的なピペッティングカラーインジケーターにより、目視による人為的なピペッティングエラーを防止します。
LNA 設計のエキスパート
20年にわたるLNA 設計の経験により、50を超えるさまざまなパラメーターを組み込んだLNA 設計アルゴリズムを開発・最適化をしました。そして厳しい性能基準に対して徹底的にラボ検証がなされました。その結果、優れたターゲット検出をお約束するアッセイが完成しました。転写産物ごとに最大3つの異なるアッセイ設計を選択し、一般的なmRNA およびlncRNA に対するアッセイをすべてウェットラボで検証しました。 あとはQuantiNova LNA プローブ PCR アッセイを混ぜるだけです。最適化に時間や費用はもう必要はありません。
迅速かつ簡便なワークフロー
迅速かつ簡単な2時間のワークフローは、時間と労力を節約し、自動化処理にも適しています。 シンプルなプロトコールは、いずれのqPCR装置とも互換性があり(図 QuantiNova LNA Probe PCR Assays deliver consistent performance across all thermal cycler instruments 参照)。さらに、RT ステップで生成されたcDNA はQuantiNova LNA プローブPCR システム全体で使用できるため、研究ニーズに応じてアッセイからパネルへのシームレスな移行が可能になり、時間とサンプルを節約できます。
図参照
 QuantiNova LNA Probe PCR Assays enable both high-expression and low-expression detection of mRNA.
QuantiNova LNA Probe PCR Assays enable both high-expression and low-expression detection of mRNA.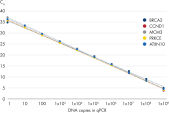 Demonstrated lot-to-lot reproducibility.
Demonstrated lot-to-lot reproducibility. Consistently high efficiencies throughout the expected GC content range of the human transcriptome.
Consistently high efficiencies throughout the expected GC content range of the human transcriptome.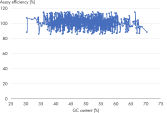 QuantiNova LNA Probe PCR Assays deliver consistent performance across all thermal cycler instruments.
QuantiNova LNA Probe PCR Assays deliver consistent performance across all thermal cycler instruments.
原理
加水分解プローブによる高いパフォーマンス
QuantiNova LNA PCRアッセイは、FAM標識の加水分解プローブによる検出を行います。これは目的のPCR産物のみを検出できるため、高い検出感度と精度の両立がなされます。プローブベースの検出は、非特異的なPCR産物やプライマーダイマーのような、競争阻害やパフォーマンスの低下につながる不適切なPCR増幅を抑制して高いPCR特異性を示すことができます。QuantiNova LNA プローブPCR システムは、すでに非特異的な増幅を回避するように最適化されていますので、安心して正確かつ高感度な検出系をご利用いただけます。
包括的なカバレッジ
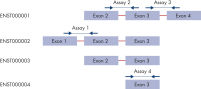
独自のアルゴリズムは130万種以上のQuantiNova LNAプローブPCRアッセイを設計し、感度、正確さに優れたmRNAおよびlncRNA解析を提供します。事前に設計されたアッセイは、Ensemblデータベース内のヒト、マウス、ラットの遺伝子のほとんどの転写産物をカバーし、PCRベースの遺伝子発現研究を高いレベルで実現します。ほとんどのアッセイは、可能な限りイントロンをまたぎ、目的のRNAのみを検出するように設計されています。イントロンをまたがないアッセイは、ターゲットに1つのエクソンがある場合などになりますが、不要なシグナルはQuantiNova Reverse Transcriptionキットに含まれるgDNA除去ステップにより容易に除去・低減できるように設計されています。
ターゲットに最適なアッセイの選択
事前に設計されたQuantiNova LNA プローブPCRアッセイを使用すれば、ヒト、マウス、ラットのmRNAまたはlncRNAを正確かつ高感度に検出できます。それは一般的な転写産物、特定の転写産物または転写産物アイソフォームを見分けた検出などを含みます。
ほとんどのヒト、マウス、ラットの遺伝子には転写産物が1つしかありませんが、複数の転写産物がある場合は、同じアルゴリズムを使用して転写産物ごとに最大3つのアッセイを設計しました。これらのアッセイのデザインとプライマーの配置は、使用する目的に合わせて異なります。弊社のアッセイ選択ガイドは、各カテゴリーから最適なアッセイをすばやく特定するのに役立ちます。ターゲットのアッセイを検索したら、結果を確認し、目的の用途に一致する推奨アッセイを確認してください。
- Best coverage: 一般的な転写産物の検出
- Best transcript assay: 特定の転写産物の検出
- Best transcript-specific assay: 特定の転写産物アイソフォームの区別して検出
詳細については、下表を参照してください。
| 遺伝子をカバーするアッセイ | 転写産物特異的なアッセイ | ||
|---|---|---|---|
| QIAGEN推奨のアッセイ | “Best coverage”表記のアッセイ | “Best transcript assay”表記のアッセイ | “Best transcript-specific assay”表記のアッセイ |
| アッセイデザインとカバレッジ | 対象遺伝子の生物学的に関連した転写産物の多くをカバー | 対象の遺伝子転写産物を特異的に検出するように最適化され、転写産物の多くのアイソフォームをカバー | 転写産物のスプライスバリアントに特異的 |
| 使用用途 | 一般的な転写産物のスクリーニングや目的の遺伝子がサンプル中で発現しているかどうかを検出するとき できるだけ多くの転写産物やアイソフォームの検出をするとき | 特異的転写産物を検出するとき | 転写産物の特異的アイソフォーム間を区別するとき |
無料のデータ解析ツール
GeneGlobeデータ解析センター の無料のWebベースのデータ解析ツールは、使いやすいウィザードが含まれており、データの標準化と解析を指示に従って選択するだけで、論文や学会発表に対応した結果の図を作成できます。
あらゆる研究のためのリファレンス遺伝子アッセイ
ヒト、マウス、ラットの幅広いリファレンス遺伝子アッセイは、高品質なデータの標準化と、信頼ある結果担保するために利用できます。これらのアッセイは、内因性のコーディングRNA、ロングノンコーディングRNA(lncRNA)、および一般に多種多様な組織で恒常的に発現される小さな核小体RNAをターゲットとしています。リファレンス遺伝子のアッセイは蛍光色素FAMで標識されていて、QuantiNova LNA PCRシステムのリファレンス遺伝子として検証され、QuantiNova RTおよびPCR試薬により適切に機能します。
mRNA / lncRNA qPCR結果の標準化
標準化により、実験における生物学的に変化に関係のない、技術的およびサンプル間での誤差を取り除くことができます。適切な標準化は、リアルタイムPCRの実験結果を正しく解析および解釈するために重要です。 一般的には、安定して発現するリファレンス遺伝子が標準化に用いられます。
実際のmRNA / lncRNA発現解析をする前に、いくつかの内因性のコントロールリファレンス遺伝子を検証することをおすすめします。これらの候補遺伝子は、一連の実験においてすべてのサンプルで安定して発現していると思われる遺伝子から選択する必要があります。それら安定して発現しているmRNAまたはlncRNAは、文献や既存データ(例えば、NGSまたはqPCRパネルスクリーニング)などでも示されています。QuantiNova LNA PCRシステムでは、安定発現する傾向にある候補遺伝子の検証済みリファレンス遺伝子アッセイを提供しています。
すべてのリファレンス遺伝子候補は、各実験系で経験的に検証する必要があります。 多くのmRNA / lncRNAをプロファイリングするときのPCRパネルを標準化する1つの選択肢として、グローバル平均、つまり、すべての発現したmRNA / lncRNAの平均に対して標準化することです。これは、高いコールレート(発現した遺伝子)のサンプルでは良い選択肢ですが、コールレートの低いサンプルでは注意して使用する必要があります。 また、一般的な遺伝子発現レベルが変化するサンプルでは、これは適切な選択肢ではありません。 標準化の詳細なガイダンスは、GeneGlobeデータ解析センターでも記載があります。
操作手順
1ステップまたは2ステップqRT-PCRの選択
QuantiNova LNAプローブPCRアッセイは、定評のあるQuantiNovaケミストリーを使用して最適化されています。QuantiNova Reverse Transcriptionキットで逆転写を行うと、最良の結果が得られ、そのcDNAはQuantiNovaプローブPCRキットのマスターミックスと、選択したQuantiNova LNAプローブPCRアッセイを組み合わせてqPCRによって定量解析を行います。
1ステップqRT-PCRの場合は、反応条件をさらに最適化する必要がないQuantiNovaプローブRT-PCRキットがおすすめです。プライマー、プローブ、RNAテンプレート、RT-MixをRT-PCRマスターミックスに添加するだけで、反応を始められます。
出荷と配送
QuantiNova LNA プローブPCRアッセイは、室温で出荷されます。在庫にあるアッセイならば数日のうちにお届けしますが、初めてのご注文の製品は、お届けまでお時間を頂戴することが予想されます。
QuantiNova LNAプローブPCRシステムを始めるのに必要なもの
Reverse transcription: QuantiNova Reverse Transcription キット
qPCR mastermix: QuantiNova プローブPCR キット
One-Step qRT-PCR (オプション): QuantiNova SYBR® Green RT-PCR キット
アッセイまたはパネル
- QuantiNova LNA Probe PCR Assays
- QuantiNova LNA Probe PCR Reference Assays
- QuantiNova LNA Probe PCR Custom Assays
- QuantiNova LNA Probe PCR Focus Panels
- QuantiNova LNA Probe PCR lncRNA Focus Panels
- QuantiNova LNA Probe PCR Custom Panels
- QuantiNova LNA Probe PCR Flexible Panels
アプリケーション
QuantiNova LNAプローブPCRアッセイは、次のようなアプリケーションに非常に適しています:
- mRNAおよびlncRNA発現分析、プロファイリングおよび定量化
- RNA-seq遺伝子発現データの検証
- 遺伝子発現プロファイリング
- シグナルとパスウェイの解析
- LNA GapmeRまたはsiRNAによる遺伝子発現ノックダウンの確認
- 疾患関連のバイオマーカーのスクリーニング、同定、検証を含むバイオマーカーの開発
- 遺伝子発現に関連する表現型の変化のモニタリング
裏付けデータと数値
Ultrafast one- or two-step procedure.