Taq PCR Master Mix Kit
簡便な PCR セットアップ用マスターミックス
簡便な PCR セットアップ用マスターミックス
Cat. No. / ID: 201445
濃縮:5 units/µl
組み換え酵素:はい
基質類似体: dNTP、ddNTP、dUTP、ビオチン-11-dUTP、DIG-11-dUTP、蛍光-dNTP/ddNTP
エクステンション率72℃ で2~4 kb/分
半減期:97℃で10分、94℃で60分
増幅効率:≥105 倍
5'->3' エキソヌクレアーゼ活性:あり
余分なA付加:あり
3'->5' エキソヌクレアーゼ活性:なし
混入しているヌクレアーゼ:なし
混入しているRNase:なし
混入しているプロテアーゼ:なし
自己プライミング活性:なし
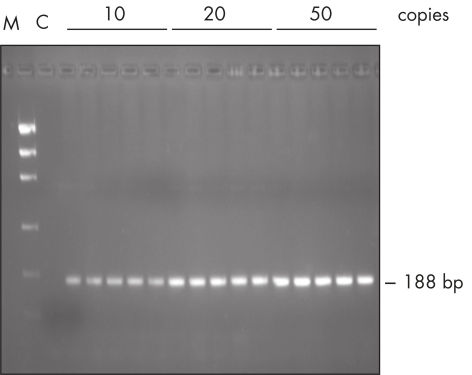
TaqPCR Master Mix Kitには 、QIAGENのTaq DNA Polymerase が、事前に混合された形で含まれています。この溶液は、QIAGEN PCR Buffer、MgCl2、および超高純度のdNTPを最適な濃度で含んでいるため、即使用することができます。PCRをセットアップするのに、プライマーとテンプレートDNAを加えるだけです。簡便なマスターミックスフォーマットにより、ピペットの誤操作が最小限に抑えられて、再現性の高いPCR結果が確実に得られます(図" 再現性のあるPCR")。Taq PCR Master Mix は最長2カ月間、2~8℃で保存することもでき、使用する場合は解凍する必要がないのでPCR セットアップがより速く行なえます。
Taq DNA Polymerase は、スタンダードなPCRから特殊なPCRまで適応できる 高品質の組み換え酵素です(図 " Tm 値の異なるプライマーへの適応性" と" 長いPCR産物の特異的増幅")。
革新的なQIAGEN PCR Bufferは、PCR至適化の操作を軽減することにより、時間と労力を節約するために開発されました。QIAGEN PCR Bufferは、KCl も (NH4)2SO4 も含んでいます(図" プライマーのアニーリングにおける特異性が増加")。このユニークなバッファーによって、特異的PCRの産物を増幅しやすくなります。PCR サイクルごとのアニーリングの間、非特異的なプライマー結合に対する特異的な結合の比率が、バッファーによって高く保たれます。このPCRバッファーは、KClと (NH4)2SO4 のユニークな配合比により、従来のPCR バッファーに比べ幅広いアニーリング温度やMg2+濃度の範囲で厳密なプライマーアニーリング条件を実現します。異なるアニーリング温度あるいはMg2+ 濃度を用いて行なうPCRの至適化は、劇的に緩和され、多くの場合、不要となります(図 " 幅広い至適アニーリング温度"および" 異なったマグネシウム濃度への適応 ")。
TaqPCR Master Mix Kitは、以下のようなスタンダードな用途にも特殊な用途にも使用できます。
| Features | Specifications |
|---|---|
| Applications | PCR, RT-PCR, DNA fingerprinting |
| dNTP's included | Yes (in Master Mix) |
| Mastermix | Yes |
| Reaction type | PCR amplification |
| Enzyme activity | 5' -> 3' exonuclease activity |
| Real-time or endpoint | Endpoint |
| Sample/target type | Genomic DNA and cDNA |
| Single or multiplex | Single |
| With/without hotstart | Without hotstart |