dPCR LNA Mutation Assay (200)
Cat. No. / ID: 250200
Caractéristiques
- La conception duplex des dosages permet de détecter les séquences mutées et de type sauvage
- Choix entre les combinaisons de colorants fluorescents FAM + HEX ou Atto 550 + ROX
- Les amorces et sondes aux performances améliorées par des LNA accroissent la spécificité et la sensibilité des dosages
- Dosages testés par dPCR en laboratoire humide, disponibles pour plus de 200 cibles
- Adaptés à une utilisation avec l’ADN tumoral circulant, les biopsies liquides, les tissus FFPE et autres échantillons de tissus
- Conçus pour une utilisation avec les QIAcuity Probe PCR Kits
Détails produit
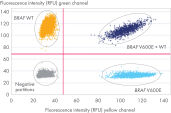
Les dPCR LNA Mutation Assays permettent la détection de mutations de séquences individuelles sélectionnées dans de vastes bases de données validées telles que COSMIC. Les conceptions des dosages ont été testées par dPCR en laboratoire humide, et les données obtenues sont disponibles. La sensibilité de la détection de la mutation cible est de 0,1 % sur un fond de type sauvage dans un puits de QIAcuity Nanoplate. Une sensibilité encore plus élevée peut être obtenue en divisant la réaction entre plusieurs puits et en combinant les analyses. La possibilité de choisir entre deux combinaisons de colorants différentes permet de détecter la séquence mutante et de type sauvage, ainsi que d’effectuer une analyse multiplex de deux mutations cibles dans un même puits.
Les dosages sont disponibles au format tube sous forme de dosages prêts à l’emploi à la concentration 30x, comprenant deux amorces et deux sondes pour la séquence mutante et de type sauvage. Les dPCR LNA Mutation Assays doivent être utilisés avec le QIAcuity Probe PCR Kit.
Performances
La dPCR offre une meilleure sensibilité dans la détection et la quantification absolue des événements rares/variants de séquence, rendant possible la détection de la présence d’une mutation à 0,1 % sur un fond d’ADN génomique de type sauvage. Le partitionnement permet d’augmenter la concentration spécifique de la cible. L’amplification et la détection en point final au-dessus du bruit, avec un appel de cible positif/négatif binaire permet une quantification précise, qui est indépendante des performances d’amplification (liées par exemple à la présence d’inhibiteurs de la PCR).
Principe
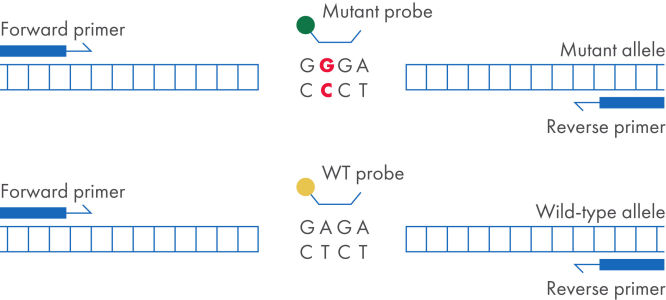
Les dPCR LNA Mutation Assays sont des dosages duplex à base de sonde d’hydrolyse, qui permettent de déterminer la présence de séquences de mutation spécifiques. Une sonde détecte l’allèle mutant et l’autre détecte l’allèle de type sauvage. La PCR numérique constitue la méthode la plus sensible et la plus fiable pour détecter une séquence d’ADN mutante sur un fond de type sauvage. Les dPCR LNA Mutation Assays font appel à des amorces et sondes améliorées par des LNA qui accroissent la spécificité et la sensibilité des dosages, rendant possible la détection d’une mutation présente à 0,1 % sur un fond d’ADN génomique de type sauvage dans un même puits de nanoplaque.
Deux options de détection de fluorescence permettent une analyse multiplex de deux cibles de mutation dans un puits. Pendant la configuration du produit, il est possible de choisir entre FAM + HEX et Atto 550 + ROX pour la détection de la séquence mutante et de type sauvage.
Procédure
Pour configurer l’expérience de dPCR, ajouter une aliquote de l’échantillon d’ADN génomique (isolé à partir d’échantillons frais, congelés ou fixés) au mélange principal du QIAcuity Probe PCR Kit et au dPCR LNA Mutation PCR Assay. Chaque réaction est analysée dans un ou plusieurs puits de la QIAcuity Nanoplate.
Après chargement et scellage de la nanoplaque, sélectionnez le programme de thermocyclage recommandé sur l’instrument QIAcuity. Dans le type d’analyse de quantification absolue de la QIAcuity Software Suite, le résultat est indiqué en copies par microlitre. L’abondance fractionnelle calculée de la cible mutante et les figures des données correspondantes sont disponibles dans le type d’analyse Analyse des mutations.
Applications
Les dPCR LNA Mutation PCR Assays sont particulièrement adaptés à l’identification rapide et exacte de mutations de séquences spécifiques individuelles présentes dans l’ADN d’échantillons frais, congelés ou fixés.
Données et illustrations utiles
Configuration du dPCR LNA Mutation Assay.