✓ Traitement automatique des commandes en ligne 24 h/24 7 j/7
✓ Assistance technique et produits pertinente et professionnelle
✓ Commande (ou réapprovisionnement) rapide et fiable
MinElute Reaction Cleanup Kit (50)
Cat. No. / ID: 28204
✓ Traitement automatique des commandes en ligne 24 h/24 7 j/7
✓ Assistance technique et produits pertinente et professionnelle
✓ Commande (ou réapprovisionnement) rapide et fiable
Caractéristiques
- Très faibles volumes d’élution
- Procédure rapide et manipulation facile
- Récupérations importantes et reproductibles
- Colorant de chargement gélifié pour simplifier l’analyse d’échantillon
Détails produit
Le MinElute Reaction Cleanup Kit contient des colonnes de centrifugation, tampons et tubes de prélèvement pour la purification sur membrane de silice d’ADN de 70 pb à 4 kb à partir de réactions enzymatiques. Les colonnes de centrifugation sont conçues pour permettre l’élution dans de très faibles volumes (de l’ordre de 10 µl), ce qui donne des rendements élevés d’ADN hautement concentré. Un indicateur de pH intégré simplifie la détermination du pH optimal pour la liaison de l’ADN à la colonne de centrifugation. La procédure peut être entièrement automatisée sur le système QIAcube Connect. Les fragments d’ADN purifiés avec le système MinElute peuvent s’utiliser directement dans toutes les applications, y compris le séquençage, l’analyse de puce à ADN, la ligature et la transformation, la digestion par enzymes de restriction, le marquage, la micro-injection, la PCR et la transcription in vitro.
Pour des résultats optimaux, il est recommandé d’utiliser ce produit avec le QIAvac 24 Plus.
Performances
Le MinElute Reaction Cleanup Kit permet le nettoyage d’ADN allant jusqu’à 5 µg (70 pb à 4 kb) à partir de réactions enzymatiques, ce qui donne des rendements élevés d’ADN adapté à de nombreuses applications. Le kit contient des colonnes de centrifugation destinées au nettoyage de réactions enzymatiques. À l’aide d’une microcentrifugeuse ou d’une rampe à vide, vous obtenez rapidement une grande concentration de fragments d’ADN (70 pb à 4 kb). (Les fragments d’ADN supérieurs à 4 kb doivent être purifiés avec le système QIAquick.)
| Protéine | Poids moléculaire par sous-unité enzymatique (kDa) |
|---|---|
| ADN polymérase I | 109 |
| Fragment de Klenow | 62 |
| Phosphatase alcaline intestinale de veau | 69 |
| ADN ligase T4 | 55 |
| Polynucléotide kinase T4 | 35 |
| Transférase terminale | 32 |
| DNase I | 31 |
| Enzymes de restriction | Variable |
Principe
Les MinElute Kits contiennent une membrane de silice permettant la liaison de l’ADN dans un tampon de forte salinité et l’élution avec un tampon de faible salinité ou de l’eau. La procédure de purification permet d’éliminer les amorces, les nucléotides, les enzymes, l’huile minérale, les sels, l’agarose, le bromure d’éthidium et d’autres impuretés des échantillons d’ADN. La technologie à membrane de silice permet d’écarter les problèmes liés aux résines et aux bouillies non homogènes. Des tampons de liaison spéciaux sont optimisés pour des applications spécifiques et favorisent l’adsorption sélective de molécules d’ADN de tailles bien précises.
Colorant de chargement gélifié
Afin de faciliter un traitement et une analyse plus rapides et pratiques des échantillons, un colorant de chargement gélifié est fourni. Le colorant de chargement GelPilot contient trois colorants de suivi (xylène cyanol, bleu de bromophénol et orange G) pour faciliter l’optimisation du temps d’exécution du gel d’agarose et empêcher les petits fragments d’ADN de migrer trop loin (voir l’illustration « Colorant de chargement GelPilot »).
Voir les illustrations
Procédure
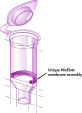
Le système MinElute utilise une procédure simple de liaison-lavage-élution (voir le schéma « Procédure MinElute »). Le tampon de liaison est ajouté directement à la réaction enzymatique puis le mélange est appliqué à la MinElute Spin Column. Le tampon de liaison contient un indicateur de pH, cela simplifie la détermination du pH optimal pour la liaison de l’ADN (voir l’illustration « Colorant indicateur de pH »). Les acides nucléiques se fixent à la membrane de silice dans les conditions de forte salinité du tampon. Les impuretés sont éliminées et l’ADN pur est élué dans un petit volume de tampon de faible salinité ou d’eau, prêt à être utilisé dans d’autres applications.
Manipulation
Les MinElute Spin Columns offrent deux possibilités de manipulation pratiques (voir le schéma « Procédure MinElute »). Les colonnes de centrifugation peuvent être insérées dans une microcentrifugeuse de paillasse classique ou dans n’importe quelle rampe à vide dotée de connecteurs Luer, de type QIAvac 24 Plus ou QIAvac 6S avec QIAvac Luer Adapters. Le MinElute Reaction Cleanup Kit, tout comme d’autres kits QIAGEN avec colonnes de centrifugation, peut être entièrement automatisé sur le QIAcube Connect, cela permet d’accroître la productivité et de standardiser les résultats (voir les illustrations « Possibilités de manipulation des colonnes de centrifugation A, B, C, D et E » et « QIAcube Connect »).
Voir les illustrations
 Colorant indicateur de pH.
Colorant indicateur de pH.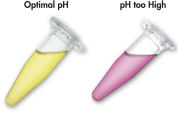 Possibilités de manipulation des colonnes de centrifugation – A.
Possibilités de manipulation des colonnes de centrifugation – A. Possibilités de manipulation des colonnes de centrifugation – B.
Possibilités de manipulation des colonnes de centrifugation – B. Possibilités de manipulation des colonnes de centrifugation – C.
Possibilités de manipulation des colonnes de centrifugation – C.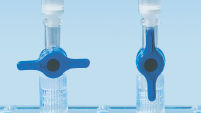 Possibilités de manipulation des colonnes de centrifugation – D.
Possibilités de manipulation des colonnes de centrifugation – D. Possibilités de manipulation des colonnes de centrifugation – E.
Possibilités de manipulation des colonnes de centrifugation – E. QIAcube Connect.
QIAcube Connect.
Applications
Les fragments d’ADN purifiés à l’aide du système MinElute peuvent s’utiliser directement dans toutes les applications, notamment :
- Séquençage
- Analyse de puce à ADN
- Ligature et transformation
- Digestion par enzymes de restriction
- Marquage
Données et illustrations utiles
Possibilités de manipulation des colonnes de centrifugation – E.

Specifications
| Features | Specifications |
|---|---|
| Binding capacity | 5 µg |
| Fragment size | 70 pb – 4 kb |
| Processing | Manual |
| Elution volume | 10 µl |
| Recovery: oligonucleotides dsDNA | Récupération : oligonucléotides, ADNdb |
| Removal <10mers 17–40mers dye terminator proteins | Élimination < 40 mères |
| Format | Tube |
| Sample type: applications | ADN, oligonucléotides : réactions enzymatiques |
| Technology | Technologie à base de silice |