✓ Traitement automatique des commandes en ligne 24 h/24 7 j/7
✓ Assistance technique et produits pertinente et professionnelle
✓ Commande (ou réapprovisionnement) rapide et fiable
QIAquick 96 PCR Purification Kit (4)
Cat. No. / ID: 28181
✓ Traitement automatique des commandes en ligne 24 h/24 7 j/7
✓ Assistance technique et produits pertinente et professionnelle
✓ Commande (ou réapprovisionnement) rapide et fiable
Caractéristiques
- Jusqu’à 95 % de récupération d’ADN prêt à l’emploi
- Procédure rapide et pratique
- Nettoyage d’ADN jusqu’à 10 kb en trois étapes simples
Détails produit
Les QIAquick 96 PCR Purification Kits contiennent des plaques à 96 puits, tampons et tubes de prélèvement pour la purification à haut débit sur membrane de silice de produits de PCR > 100 pb. L’ADN jusqu’à 10 kb est purifié suivant une procédure simple et rapide de liaison–lavage–élution, avec un volume d’élution de 60 à 80 µl (qui donne un volume d’éluat de 40 à 60 µl). La procédure de nettoyage peut être entièrement automatisée sur la station de travail universelle BioRobot à l’aide du QIAquick 96 PCR BioRobot Kit.
Performances
Voir les illustrations
Principe
Les QIAquick 96 Kits contiennent une membrane de silice permettant la liaison de l’ADN dans un tampon de forte salinité et l’élution avec un tampon de faible salinité ou de l’eau. La procédure de purification permet d’éliminer les amorces, les nucléotides, les enzymes, l’huile minérale, les sels, l’agarose, le bromure d’éthidium et d’autres impuretés des échantillons d’ADN. La technologie à membrane de silice permet d’écarter les problèmes liés aux résines et aux bouillies non homogènes. Des tampons de liaison spéciaux sont optimisés pour des applications spécifiques et favorisent l’adsorption sélective de molécules d’ADN de tailles bien précises.
La procédure QIAquick 96 permet la purification en parallèle de 96 échantillons de PCR maximum par purification sous vide sur un QIAvac 96.
Le QIAquick 96 PCR BioRobot Kit est un kit spécifique optimisé pour être utilisé sur le système universel BioRobot. Ce kit propose des modules QIAquick 96 ainsi que tous les tampons et éléments en plastique nécessaires au nettoyage automatisé à haut débit de 96 échantillons de PCR.
Procédure
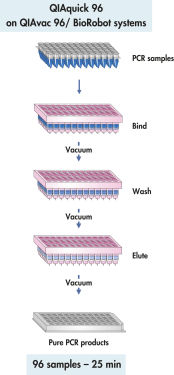
Le système QIAquick utilise une procédure simple de liaison–lavage–élution (voir le schéma « Procédure QIAquick 96 »). Le tampon de liaison est ajouté directement à l’échantillon de PCR ou à la réaction enzymatique, puis le mélange est appliqué à la plaque à 96 puits. Les acides nucléiques se fixent à la membrane de silice dans les conditions de forte salinité du tampon. Les impuretés sont éliminées et l’ADN pur est élué dans un petit volume de tampon de faible salinité ou d’eau, prêt à être utilisé dans d’autres applications.
Manipulation
Les modules à plusieurs puits QIAquick sont traités par purification sous vide sur les rampes à vide QIAvac. Le QIAquick 96 PCR Purification Kit implique d’utiliser le QIAvac 96 Vacuum Manifold. La procédure de nettoyage peut être entièrement automatisée sur les stations de travail universelles BioRobot à l’aide du QIAquick 96 PCR BioRobot Kit.
Voir les illustrations
Applications
Les fragments d’ADN purifiés avec le système MinElute ou QIAquick peuvent s’utiliser directement dans diverses applications, y compris le séquençage, l’analyse de puce à ADN, la ligature et la transformation, la digestion par enzymes de restriction, le marquage, la micro-injection, la PCR et la transcription in vitro.
Données et illustrations utiles
Procédure QIAquick 96.
Le QIAquick 96 Kit utilise une procédure de liaison-lavage-élution avec le QIAvac 96 ou le système universel BioRobot.
Specifications
| Features | Specifications |
|---|---|
| Binding capacity | 10 µg |
| Processing | Manuel/Automatisé |
| Removal <10mers 17–40mers dye terminator proteins | Élimination < 40 mères |
| Sample type: applications | ADN, oligonucléotides : réactions de PCR |
| Format | Plaque de 96 puits |
| Fragment size | 100 pb – 10 kb |
| Technology | Technologie à base de silice |
| Recovery: oligonucleotides dsDNA | Récupération : oligonucléotides, ADNdb |
| Elution volume | 60 – 80 µl |