Products
Caractéristiques
- Dosages haute performance créés à l’aide d’un outil de conception pratique et du même algorithme utilisé pour nos dosages préconçus
- Dosages personnalisés pour correspondre exactement à votre cible et répondre à vos besoins, y compris pour la détection de nouveaux transcrits, d’une isoforme spécifique ou d’un variant d’épissage
- Possibilité de soumettre jusqu’à 10 séquences et de concevoir des dosages pour différencier ou détecter toutes les séquences
- Courtes amorces améliorées par des LNA avec une haute spécificité et une plus grande flexibilité dans le positionnement du dosage
- Quantification absolue des changements d’expression avec la dPCR à l’aide de l’instrument QIAcuity et du QIAcuity EG PCR Kit
Détails produit
Les QuantiNova LNA PCR Custom Assays permettent d’analyser avec exactitude et sensibilité l’expression génique de tout ARN cible à l’aide de la PCR digitale à base d’EvaGreen améliorée par des LNA. Notre solide algorithme de conception couvre toute la base de données Ensembl pour les cibles chez l’homme, la souris et le rat, et il simplifie la création des dosages. Les dosages personnalisés sont parfaitement adaptés aux cibles pour lesquelles aucun dosage préconçu n’est disponible, pour la discrimination des variants d’épissage et pour la vérification de nouveaux ARNm ou lncARN. Pour la PCR digitale, nous avons optimisé les dosages avec les QIAcuity EG PCR Kits et l’instrument QIAcuity et créé une méthode de quantification absolue simple et rapide qui prend seulement 2 heures.
Avez-vous l’intention d’utiliser des QuantiNova LNA PCR Custom Assays pour l’analyse par qPCR ? Vous trouverez des détails sur les produits spécifiques à la qPCR et sur leurs performances dans la page de catalogue consacrée à la qPCR.
Vous avez besoin d’un devis pour votre projet de recherche ou vous souhaitez discuter de votre projet avec notre équipe de spécialistes ? Il vous suffit de nous contacter !
Performances
Analyse de PCR digitale optimisée avec les QIAcuity EG PCR Kits et l’instrument QIAcuity
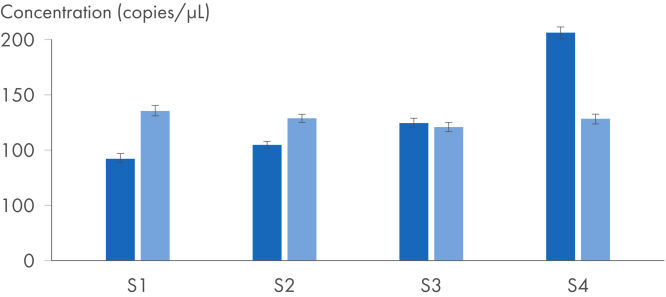
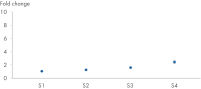
Les QuantiNova LNA PCR Assays ont été développés pour permettre une amplification des cibles avec une grande spécificité. Cela peut être quantifié par deux méthodes de PCR : la qPCR avec des thermocycleurs en temps réel classiques et la PCR digitale à l’aide de l’instrument QIAcuity et du QIAcuity EG PCR Kit (voir figure « Utilisation des QuantiNova LNA PCR Assays pour la PCR digitale »). Après la synthèse de l’ADNc à l’aide du QuantiTect Reverse Transcription Kit, la PCR digitale permet une quantification des cibles très précise, assurant la détection d’infimes changements d’expression aux concentrations les plus faibles. Un sous-ensemble des principaux dosages a été validé expérimentalement sur l’instrument QIAcuity.
Dosages hautement sensibles et spécifiques améliorés par des LNA
Les QuantiNova LNA PCR Custom Assays sont créés à l’aide de critères de conception rigoureux et d’algorithmes validés en laboratoire pour assurer une très grande spécificité et efficacité et produire des résultats fiables et exacts dans l’analyse de l’expression génique.
Comme la haute affinité de liaison des bases des LNA augmente la flexibilité du placement des amorces sur le transcrit, nous pouvons utiliser un positionnement judicieux afin de concevoir des dosages pour les cibles habituellement difficiles à analyser. Cela vous procure une meilleure discrimination des cibles et une quantification fiable et robuste, même pour les cibles riches en AU, les transcrits en faible abondance, les cibles avec une teneur élevée en structure secondaire et les échantillons très complexes.
Cette haute sensibilité ’des dosages garantit d’excellentes efficacités d’amplification à partir de 1 molécule d’ARN, simplifiant la détection des cibles en faible abondance telles que les lncARN avec de plus faibles quantités de matériel de départ. La spécificité augmentée par le positionnement judicieux des LNA vous fait profiter d’un plus grand rapport signal sur bruit, permettant la discrimination des séquences qui ne diffèrent que par un seul nucléotide et éliminant l’amplification non spécifique et la formation de dimères d’amorces.
Créés à l’aide d’un algorithme de conception de LNA sophistiqué
Grâce à nos 20 ans d’expérience dans la conception de LNA, nous avons pu développer et optimiser notre algorithme de conception de LNA sophistiqué, qui intègre plus de 50 paramètres différents – tous complètement validés selon des critères de performances rigoureux –, afin d’assurer un dosage optimal pour détecter les cibles. Cet algorithme exclusif a été utilisé pour concevoir plus de 1,3 million de dosages et est disponible pour satisfaire à vos demandes spécifiques par l’intermédiaire de notre outil pratique de création de dosages personnalisés dans GeneGlobe.
Principe
PCR digitale à base d’EvaGreen
Pour une quantification absolue par dPCR, utilisez les QuantiNova LNA PCR Assays avec le QIAcuity EG PCR Kit, qui fait appel une détection de fluorescence à base d’EvaGreen. EvaGreen est un colorant intercalant qui se fixe sur l’ADN double brin (comme SYBR® Green) et émet de la fluorescence après sa fixation sur l’ADN. L’utilisation de la dPCR à base d’EvaGreen offre commodité et économies, parce que vous n’avez besoin que du jeu d’amorces d’amplification et de détection du produit. Elle assure aussi une résolution plus élevée dans la réaction de dPCR, qui est divisée en milliers de partitions sur la nanoplaque de dPCR, de sorte que la concentration en amorces nécessaire pour la réaction de dPCR est moitié moindre que pour la qPCR.
Le processus de conception personnalisée
L’outil de conception de QuantiNova LNA PCR Custom Assay vous permet de concevoir facilement des dosages de PCR améliorés par des LNA hautement sensibles et spécifiques pour tout ARNm ou lncARN non couvert par un dosage préconçu. À l’aide de l’algorithme avancé de conception de QuantiNova PCR Assays, de nombreuses combinaisons de dosages sont évaluées en fonction de plus de 50 critères différents afin de trouver le dosage optimal pour votre cible en quelques minutes. Cet outil a été conçu pour les cibles d’ARNm et de lncARN de l’homme, de la souris et du rat d’au moins 55 nucléotides et procède à des blasts dans les bases de données pertinentes pour l’espèce concernée, dont Ensembl et RefSeq. Pour les cibles géniques connues, les dosages sont conçus par défaut avec un chevauchement d’intron, comme les dosages préconçus, pour prévenir le risque d’amplification de l’ADNg.
Options avancées exclusives pour la conception des dosages
L’outil de conception de dosages personnalisés comporte des options pour des conceptions spécifiques à un transcrit pour la discrimination des variants d’épissage, des SNP ou des isoformes. Il est aussi possible de concevoir des dosages communs à tous les variants de transcription. Vous pouvez soumettre 10 séquences cibles différentes à la fois et concevoir 10 dosages différents ou concevoir un dosage commun pour tous les transcrits correspondants.
Les Reference Gene Assays peuvent être facilement combinés avec les dosages personnalisés
Une large sélection de Reference Gene Assays, validés fonctionnellement pour l’homme, la souris et le rat, est disponible pour permettre une normalisation des données de haute qualité et assurer des résultats fiables. Ces dosages ciblent des ARN codants, de longs ARN non codants et de petits ARN nucléolaires endogènes qui sont généralement exprimés constitutivement dans une grande variété de tissus.
Normalisation des résultats de qPCR d’ARNm/lncARN
La normalisation élimine les variations techniques et biologiques entre les échantillons sans lien avec les changements biologiques étudiés. Il est essentiel d’effectuer une normalisation appropriée pour garantir une analyse et une interprétation correctes des résultats. Le plus souvent, on utilise des gènes de référence exprimés de façon stable pour la normalisation.
Il est généralement recommandé de tester plusieurs gènes de référence de contrôle endogène avant de configurer votre analyse réelle d’expression des ARNm/lncARN. Ces gènes candidats doivent être choisis parmi des gènes qui doivent être exprimés de façon stable sur toute la gamme d’échantillons étudiés. Il peut s’agir d’ARNm ou de lncARN exprimés de façon stable sélectionnés en fonction de la littérature ou de données préexistantes (p. ex. criblage de panel de qPCR ou NGS). Le système QuantiNova LNA PCR comporte des dosages de gènes de référence validés pour les ARN qui ont tendance à être exprimés de façon stable et sont donc de bons candidats comme gènes de référence.
Tous les gènes candidats doivent être validés empiriquement pour chaque étude. Une façon de normaliser un panel de PCR lors du profilage d’un grand nombre d’ARNm/lncARN consiste à effectuer la normalisation par rapport à la moyenne globale – la moyenne de tous les ARNm/lncARN exprimés. Cette approche peut convenir aux échantillons à fort taux d’appel (gènes exprimés), mais elle doit être utilisée avec précaution pour les échantillons à faible taux d’appel. Cette approche n’est pas non plus appropriée pour les échantillons dans lesquels le niveau d’expression génique global est modifié. Vous trouverez d’autres conseils de normalisation dans le GeneGlobe Data Analysis Center.
Analyse des données de PCR digitale
La QIAcuity Software Suite est utilisée pour analyser les données de dPCR et comprend un test d’expression génique, qui donne les résultats sous forme d’ampleur du changement et d’ampleur de la régulation avec des figures prêtes pour la publication.
Procédure
RT-dPCR en deux étapes
La réalisation de la réaction de transcription inverse à l’aide du QuantiTect Reverse Transcription Kit (réf. 205311, 205313, 205314) donne les meilleurs résultats. Il n’est pas recommandé d’utiliser le QuantiNova Reverse Transcription Kit. L’ADNc obtenu est ensuite quantifié par dPCR à l’aide des mélanges principaux du QIAcuity EG PCR Kit associé avec le QuantiNova LNA PCR Assay choisi.
Remarque importante : Les quantités réactionnelles indiquées entre parenthèses après les noms des produits de dosage sont destinées à une utilisation en qPCR. La PCR digitale ne requiert qu’une concentration moitié moindre en amorces, mais les volumes réactionnels sont différents. Avec la QIAcuity Nanoplate 26K, vous pouvez configurer les nombres de réactions indiqués ; avec la Nanoplate 8.5K vous pouvez en configurer 3,3 fois plus. Reportez-vous au manuel pour des instructions d’utilisation de QIAcuity en dPCR.
Expédition et livraison
Les QuantiNova LNA PCR Assays sont expédiés à température ambiante. Les dosages en stock sont livrés en 1–5 jours. Pendant la période d’accès anticipé, les délais de livraison peuvent être plus longs.
Ce dont vous avez besoin pour commencer la PCR digitale avec le système QuantiNova LNA PCR
Transcription inverse : QuantiTect Reverse Transcription Kit (réf. 205311, 205313, 205314)
Mélange principal de dPCR : QIAcuity EG PCR Kit
Dosages :
Applications
Les QuantiNova LNA PCR Custom Assays sont parfaitement adaptés aux applications telles que :
- L’analyse d’expression, le profilage et la quantification des ARNm et lncARN
- La validation des données d’expression génique RNA-seq
- Le profilage d’expression génique
- L’analyse des signaux et des voies
- La confirmation de l’inhibition de l’expression génique par des LNA GapmeRs ou des pARNi
- Le développement de biomarqueurs, y compris le criblage, l’identification et la validation de biomarqueurs associés à une maladie
- Le suivi des changements phénotypiques en lien avec l’expression génique
Il est également possible d’utiliser plusieurs QuantiNova LNA PCR Assays pour examiner un panel de gènes spécifique.
Données et illustrations utiles
Analyse de l’expression du gène IL-4 – détection de faibles changements d’expression avec la plus grande précision.