✓ Procesamiento automático sin interrupción de pedidos en línea
✓ Servicio técnico y para productos experto y profesional
✓ Realización y repetición de pedidos rápidas y fiables
QIAEX II Gel Extraction Kit (150)
Cat. No. / ID: 20021
✓ Procesamiento automático sin interrupción de pedidos en línea
✓ Servicio técnico y para productos experto y profesional
✓ Realización y repetición de pedidos rápidas y fiables
Características
- Extracción eficiente de ADN de entre 40 bp y 50 kb
- Extracción en gel a partir de geles de agarosa con TAE o TBE y geles de poliacrilamida
- Sin yoduro de sodio que interfiera con las reacciones posteriores
- Sin cizallamiento de fragmentos grandes de ADN
Detalles del producto
El sistema QIAEX II incluye una suspensión de las partículas de sílice a las que los fragmentos de ADN se unen en presencia de sales caotrópicas. La suspensión QIAEX II se añade a soluciones o cortes de gel de agarosa solubilizada y une el ADN. Las partículas se recogen mediante una breve centrifugación, se lavan y el ADN de entre 40 bp y 50 kb se eluye en tampón Tris o agua.
Rendimiento
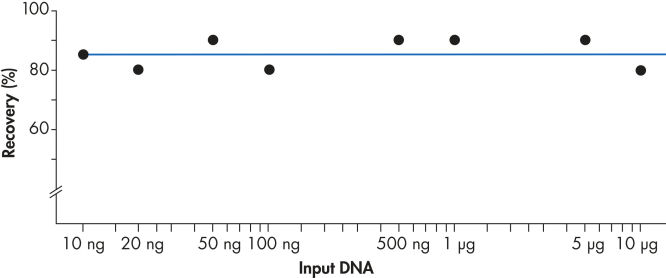
Con el uso del sistema QIAEX II, se recupera el ADN de entre 10 ng y 10 µg de forma eficiente (consulte la figura «Recuperación uniforme»). El versátil procedimiento para la purificación por lotes de los fragmentos en gel se puede escalar fácilmente a una capacidad de unión de 15 µg empleando 30 µl de suspensión QIAEX II.
El sistema QIAEX II incluye partículas de sílice para purificar fragmentos de ADN al 60–95 % (40 bp–50 kb). Un volumen de 10 µl de suspensión QIAEX II une hasta 5 µg de ADN, que posteriormente se eluye en 20 µl.
Recuperación según el tamaño
| Tamaño de ADN | Recuperación, porcentaje* |
|---|---|
| 44 bp | 75 |
| 75 bp | 75 |
| 500 bp | 95 |
| 7,5 kb | 85 |
| 23,5 kb | 75 |
| 48,5 kb | 60 |
Ver figuras
Principio
La purificación de los fragmentos de ADN con el sistema QIAEX II se basa en la solubilización de la agarosa y la adsorción selectiva de ácidos nucleicos en partículas de gel de sílice de QIAEX II en la presencia de sales caotrópicas. QIAEX II separa el ADN de las sales, la agarosa, la policrilamida, los colorantes, las proteínas y los nucleótidos sin extracción fenólica ni precipitación con etanol. QIAEX II es eficiente para cualquier tipo de agarosa en tampones con TAE o TBE.
Las partículas de QIAEX II proporcionan un residuo pastoso para la extracción en gel y garantizan una eficiente recuperación sin cizallamiento, incluso para fragmentos grandes de ADN. Los tampones optimizados permiten la recuperación de ADN sin yoduro de sodio, que es difícil de eliminar de las muestras de ADN y puede afectar las reacciones posteriores.
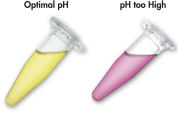
El tampón de unión y solubilización empleado con el sistema QIAEX II contiene un indicador único de pH. Con un simple cambio de color, se indica si el pH de la mezcla de unión es óptimo para la adsorción eficiente de ADN en las partículas de sílice de QIAEX II (consulte la figura «Colorante indicador de pH»). El colorante también permite la fácil visualización de agarosas no solubilizadas en la mezcla de unión, lo que garantiza que se produzca una solubilización completa para tener un máximo rendimiento.
El colorante indicador de pH en el tampón de unión y solubilización también permite que la determinación visual del pH óptimo para la adsorción de ADN (pH ≤7,5) sea sencilla. Se puede producir un pH incorrecto de mezcla de unión si el tampón de electroforesis en gel de agarosa se ha utilizado de forma frecuente o se ha preparado de forma incorrecta. En este caso, el pH se pueden ajustar fácilmente con la adición de 10 µl de acetato de sodio 3 M, con pH de 5,0.
Ver figuras
Procedimiento
Las partículas de gel de sílice de QIAEX II se añaden al corte de gel solubilizado y las partículas se recogen mediante el paso de centrifugación breve (consulte el diagrama de flujo «Procedimiento de QIAEX II»). Tras el lavado, el fragmento de ADN puro se eluye en 20 µl de tampón Tris o en agua.
El sistema QIAEX II incluye una suspensión QIAEX II junto con tampones de unión y lavado y un manual de uso integral. Se proporcionan protocolos para la purificación de ADN a partir de geles de agarosa, soluciones y geles de poliacrilamida.
Ver figuras
Aplicaciones
El ADN purificado con el sistema QIAEX II se puede utilizar directamente en la mayoría de las aplicaciones, incluyendo las indicadas a continuación:
- Digestión de restricción
- Marcado
- Ligación
- PCR
| Características | Especificaciones |
|---|---|
| Capacidad de unión | 5 µg/10 µl |
| Volumen de elución | 20 µl |
| Formato | Tubo |
| Tamaño de fragmento | 40 bp–50 kb |
| Procesamiento | Manual |
| Recuperación: ADNbc de oligonucleótidos | Recuperación: fragmentos de ADNbc |
| Eliminación, proteínas de terminador de colorante, <10 mers 17–40 mers | Eliminación, <40 mers |
| Tipo de muestra: aplicaciones | ADN: Reacciones de PCR |
| Tecnología | Tecnología de sílice |
Datos y cifras de respaldo
Recuperación uniforme.