Products
Características
- Ensayos de alto rendimiento creados con una herramienta de diseño práctica y mediante el uso del mismo algoritmo para los ensayos prediseñados
- Ensayos personalizados que coincidan con el blanco y la necesidad exacta, incluyendo la detección de nuevos transcritos o una isoforma o variante de empalme específicas
- Posibilidad de enviar hasta 10 secuencias y diseñar ensayos para diferenciar o detectar todas las secuencias.
- Cebadores breves mejorados con LNA de alta especificidad y mayor flexibilidad en la ubicación del ensayo
- Cuantificación absoluta de cambios de expresión con dPCR mediante el uso del instrumento QIAcuity y el QIAcuity EG PCR Kit
Detalles del producto
Los QuantiNova LNA PCR Custom Assays proporcionan un análisis de expresión génica exacto y sensible de cualquier blanco de ARN mediante el uso de PCR digital basada en EvaGreen y mejorada con LNA. Nuestro algoritmo de sólido diseño ofrece el respaldo de la base de datos Ensembl para blancos humanas y murinas, y agiliza la creación de ensayos. Los ensayos personalizados están pensados para blancos, las cuales no se encuentra disponible un ensayo prediseñado, para discriminar variantes de empalme y verificar ARNm o ARNInc nuevos. Para la PCR digital, hemos optimizado los ensayos con los QIAcuity EG PCR Kits y el instrumento QIAcuity, y hemos creado un flujo de trabajo de cuantificación simple, rápido y absoluto que solo supone 2 horas.
¿Planea usar los QuantiNova LNA PCR Custom Assays en el análisis de qPCR? Puede buscar detalles específicos del producto y del rendimiento de la qPCR en la página del catálogo de qPCR específica.
¿Necesita un presupuesto para su proyecto de investigación o le gustaría hablar sobre él con nuestro equipo de especialistas? ¡Solo tiene que ponerse en contacto con nosotros!
Rendimiento
Análisis de PCR optimizado con los QIAcuity EG PCR Kits y el instrumento QIAcuity
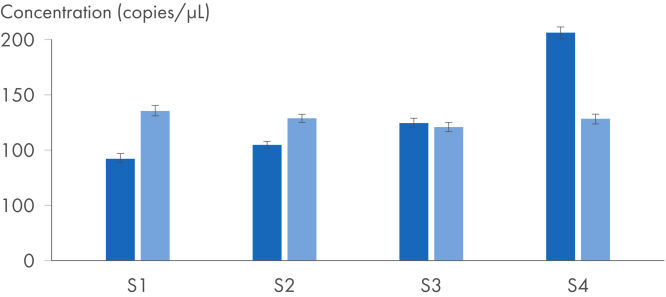
Los QuantiNova LNA PCR Assays se desarrollaron para ofrecer amplificación de blancos de alta especificidad. Esto puede cuantificarse mediante dos métodos de PCR: qPCR con termocicladores en tiempo real estándar y PCR digital con el instrumento QIAcuity y el QIAcuity EG PCR Kit (consulte la figura Uso de QuantiNova LNA PCR Assays para PCR digital). Tras la síntesis de ADNc con el QuantiTect Reverse Transcription Kit, la PCR digital ofrece cuantificación de blancos de gran precisión, que permite detectar hasta los cambios de expresión más pequeños en las concentraciones más bajas. En el instrumento QIAcuity se ha validado experimentalmente un subconjunto de ensayos clave.
Ensayos mejorados con LNA altamente sensibles y específicos
Los QuantiNova LNA PCR Custom Assays se crean a partir de criterios de diseño exigentes y algoritmos validados en laboratorio para ofrecer la máxima especificidad y eficiencia a fin de obtener resultados de análisis de expresión génica fiables y exactos.
La alta afinidad de unión de las bases de LNA aumenta la flexibilidad de ubicación del cebador en el transcrito, por lo tanto, podemos usar la ubicación inteligente para diseñar ensayos para blancos que serían difíciles de analizar de otra manera. Esto le permite realizar una mejor discriminación de blancos y una cuantificación sólida y fiable, incluso para los blancos con alto contenido de AU, los transcritos de bajos niveles de abundancia, las blancos con gran cantidad de estructura secundaria y las muestras altamente complejas.
La alta sensibilidad de los ensayos’ asegura excelentes eficiencias de amplificación de hasta 1 molécula de ARN, permitiéndole detectar fácilmente dianas de bajos niveles de abundancia como los ARNInc de menos material de partida. La mayor especificidad producto de la ubicación inteligente del LNA le proporciona una alta relación señal-ruido, que permite discriminar las secuencias que se diferencian por un solo nucleótido y eliminar la amplificación inespecífica y la formación de dímeros de cebador.
Creado con un algoritmo de sofisticado diseño de LNA
Veinte años de experiencia en el diseño de LNA nos han permitido desarrollar y optimizar nuestro sofisticado diseño de LNA, que incorpora más de 50 parámetros diferentes, todos minuciosamente validados en laboratorio con criterios de rendimiento sumamente exigentes, para garantizar un ensayo óptimo para la correcta detección de blancos. Este patentado algoritmo se ha utilizado para diseñar más de 1,3 millones de ensayos y está a disposición para sus solicitudes especiales a través de nuestro práctico creador de ensayos personalizados en GeneGlobe.
Principio
PCR digital basada en EvaGreen
Para lograr una cuantificación absoluta mediante dPCR, utilice los QuantiNova LNA PCR Assays junto con el QIAcuity EG PCR Kit, que utiliza detección de fluorescencia basada en EvaGreen. EvaGreen es un colorante intercalante que se une al ADN de doble cadena (de manera similar al SYBR® Green) y se vuelve fluorescente al unirse al ADN. El uso de dPCR basado en EvaGreen proporciona practicidad y ahorro, ya que solo necesita el juego de cebadores para amplificar y detectar el producto. También proporciona una resolución más alta en la reacción de dPCR, que se divide en miles de partes de la nanoplaca para dPCR, por lo tanto, la concentración de cebador necesaria para la reacción de dPCR es apenas la mitad de la que se necesita para qPCR.
El proceso de diseño personalizado
La herramienta de diseño QuantiNova LNA PCR Custom Assay le permite diseñar fácilmente ensayos de PCR mejorados con LNA sensibles y específicos para cualquier ARNm o ARNInc que no se encuentre disponible como ensayo prediseñado. Al utilizar el algoritmo de diseño avanzado de QuantiNova PCR Assay, se evalúan varias combinaciones de ensayos sobre la base de más de 50 criterios diferentes para encontrar el ensayo óptimo para su blanco en unos pocos minutos. La herramienta se ha diseñado para blancos de ARNm y ARNInc humano y murino de al menos 55 nucleótidos y blastocitos con las bases de datos correspondientes a las especies, entre ellas, Ensembl y RefSeq. En el caso de los genes blanco conocidos, los ensayos están diseñados como amplificación de intrones de forma predeterminada, de la misma manera que los ensayos prediseñados, para evitar el riesgo de amplificación de ADNg.
Opciones de diseño de ensayo avanzado exclusivo
La herramienta de diseño de ensayos personalizada incluye opciones para crear diseños específicos de transcritos para discriminar variantes de empalme, SNP o isoformas. Como alternativa, también se pueden crear diseños para ensayos que sean comunes a todas las variantes de transcrito. Puede enviar hasta 10 secuencias de blancos diferentes a la vez y diseñar diez ensayos diferentes o, en el caso de los transcritos relacionados, puede diseñar un ensayo en común para todos.
Los ensayos génicos de referencia pueden combinarse fácilmente con los ensayos personalizados
Se dispone de una amplia variedad de ensayos génicos de referencia humanos y murinos funcionalmente validados para permitir la normalización de datos de alta calidad y garantizar resultados fiables. Estos ensayos están dirigidos a ARN de codificación endógena, ARN extenso sin codificación y pequeñas moléculas de ARN nucleolar que generalmente se expresan constitutivamente en una amplia variedad de tejidos.
Normalización de qPCR de ARNm/ARNInc
La normalización elimina la variación técnica y biológica entre muestras que no está relacionada con los cambios biológicos en investigación. La debida normalización es fundamental para el correcto análisis e interpretación de los resultados. Lo más frecuente es que en la normalización se utilicen genes de referencia de expresión estable.
Por lo general, se recomienda analizar varios candidatos de genes de referencia de control endógeno antes de configurar el análisis de expresión de ARNm/ARNInc real. Estos candidatos deben elegirse entre genes de los que se espera una expresión estable durante todo el intervalo de muestras en investigación. Podrían ser ARNm o ARNInc de expresión estable seleccionados en función de bibliografía o datos preexistentes (p. ej., cribado de paneles de NGS o qPCR). El QuantiNova LNA PCR system ofrece ensayos de genes de referencia para ARN que suelen tener una expresión estable y, por lo tanto, son buenos candidatos como genes de referencia.
Todos los candidatos a genes de referencia deben estar empíricamente validados para cada estudio. Una opción para normalizar el panel de PCR al crear el perfil de una gran cantidad de ARNm/ARNInc es normalizar con la media global: el promedio de todos los ARNm/ARNInc expresados. Esta puede ser una buena opción en muestras con una alta tasa de éxito (genes expresados) pero debe utilizarse con cautela en muestras con bajas tasas de éxito. Tampoco es una buena opción en muestras para las cuales se modifica el nivel de expresión génica general. En GeneGlobe Data Analysis Center (Centro de análisis de datos de GeneGlobe) podrá encontrar más ayuda sobre la normalización.
Análisis de los datos de PCR digital
El QIAcuity Software Suite se utiliza para analizar los datos de dPCR e incluye una prueba de expresión génica que proporciona los resultados como múltiplo de variación y múltiplo de regulación con cifras listas para publicar.
Procedimiento
RT-dPCR de dos pasos
Los mejores resultados se obtienen cuando se lleva a cabo la reacción de transcripción inversa con el QuantiTect Reverse Transcription Kit (n.º de catálogo 205311, 205313, 205314). No es recomendable usar el QuantiNova Reverse Transcription Kit. El ADNc resultante se cuantifica mediante dPCR con las mezclas maestras del QIAcuity EG PCR Kit en combinación con su elección del QuantiNova LNA PCR Assay.
Nota importante: Las cantidades de reacciones indicadas entre paréntesis después de los nombres de producto del ensayo corresponden al uso en qPCR. La PCR digital solo requiere la mitad de la concentración de cebador, pero los volúmenes de reacción son diferentes. Si utiliza la QIAcuity Nanoplate 26K, podrá configurar la misma cantidad de reacciones que se indicaron; si utiliza Nanoplate 8.5K podrá configurar 3,3 veces más. Consulte las instrucciones para dPCR en el manual de uso de QIAcuity.
Envío y entrega
Los QuantiNova LNA PCR Assays se envían a temperatura ambiente. Los ensayos en existencias se entregan en un plazo de 1 a 5 días. Durante el periodo de acceso inicial, se prevén tiempos de entrega más prolongados.
Lo que necesita para comenzar con la PCR digital con el QuantiNova LNA PCR system
Transcripción inversa: QuantiTect Reverse Transcription Kit (n.º de catálogo 205311, 205313, 205314)
Mezcla maestra para dPCR: QIAcuity EG PCR Kit
Ensayos:
Aplicaciones
Los QuantiNova LNA PCR Custom Assays están especialmente pensados para aplicaciones como:
- Análisis de expresión de ARNm y ARNInc, creación de perfiles y cuantificación
- Validación datos de expresión génica de secuencia de ARN
- Creación de perfiles de expresión génica
- Análisis de señales y trayectorias
- Confirmación de la inactivación de la expresión génica mediante siRNA o GapmeR de LNA
- Desarrollo de biomarcadores, incluyendo el cribado, la identificación y la validación de biomarcadores asociados con enfermedades
- Supervisión de cambios fenotípicos relacionados con la expresión génica
También se pueden usar varios QuantiNova LNA PCR Assays para examinar un panel de genes específico.
Datos y cifras de respaldo
Análisis de expresión génica IL-4: detección de pequeños cambios en la expresión con máxima precisión.