✓ Procesamiento automático sin interrupción de pedidos en línea
✓ Servicio técnico y para productos experto y profesional
✓ Realización y repetición de pedidos rápidas y fiables
dPCR Microbial DNA Detection Assays
Cat. No. / ID: 250207
✓ Procesamiento automático sin interrupción de pedidos en línea
✓ Servicio técnico y para productos experto y profesional
✓ Realización y repetición de pedidos rápidas y fiables
Características
- Ensayos prediseñados para la detección de especies microbianas, genes de virulencia o genes de resistencia a antibióticos
- Ensayos para más de 700 dianas
- Herramienta personalizada de diseño de ensayos para dianas bacterianas, fúngicas o víricas disponible en GeneGlobe
- La selección de colorantes permite el multiplexado de hasta 5 dianas por reacción
- Flujo de trabajo de dPCR fácil y rápido en el QIAcuity
- Combine dianas de ADN microbiano y ARN vírico en una reacción con el uso del QIAcuity OneStep Advanced Probe Kit
Detalles del producto
La creación de perfiles y la identificación de perfiles microbianos son de interés para varios campos, como la salud humana, las pruebas de alimentos y piensos y pruebas ambientales. La identificación microbiana determina la presencia o la ausencia de un microbio en una muestra, mientras que la creación de perfiles microbianos determina su expresión relativa en dos o más condiciones experimentales, por lo tanto, necesita una muestra de referencia y un normalizador.
Nuestros dPCR Microbial DNA Detection Assays tienen como objetivo genes bacterianos, fúngicos, parasitarios, víricos, de resistencia a antibióticos o de factores de virulencia. Para las bacterias, los ensayos se dirigen al gen de ARNr 16S y, para los hongos, a los genes del ARN ribosómico. La gama consta de más de 700 ensayos distintos, 200 de los cuales están probados en laboratorios experimentales de dPCR. Para las dianas que no se incluyen en nuestra cartera de ensayos dPCR microbianos prediseñados, ofrecemos una herramienta de diseño de ensayos personalizados fácil de usar para dianas bacterianas, fúngicas y víricas.
Cada ensayo consta de un par de cebadores y una sondas de hidrólisis con un colorante fluoróforo configurable. Los fluoróforos configurables son FAM, HEX, ROX, TAMRA y Cy5 y se pueden mezclar y combinar para apoyar el análisis de hasta cinco dianas distintas en una reacción de dPCR múltiple. También ofrecemos paquetes de ensayos 5-plex de dPCR probados en laboratorios experimentales para el análisis de dianas de interés comunes; consulte las aplicaciones a continuación para obtener más detalles. Para la reacción de dPCR, el ensayo se combina con el QIAcuity Probe PCR Kit (dianas de ADN) o con el QIAcuity OneStep Advanced Probe Kit (dianas de ARN).
El ensayo funciona junto con el QIAcuity Digital PCR System y las QIAcuity Nanoplates.
¿Le gustaría obtener más información sobre este producto y que uno de nuestros especialistas en dPCR se ponga en contacto con usted? Regístrese aquí y nos pondremos en contacto con usted en breve.
Rendimiento
Detección exacta y precisa
Los dPCR Microbial DNA Detection Assays y el QIAcuity dPCR System permiten la cuantificación exacta basada en PCR digital. El análisis del material de referencia del Instituto Nacional de Normas y Tecnología (Institute of Standards and Technology, NIST) mostró la concentración esperada del molde de entrada. Consulte la figura Cuantificación de la norma de referencia 8376 del NIST con el uso del ADNg de Shigella sonnei para obtener más detalles.
Rendimiento en múltiple
La detección de dianas microbianas basada en la dPCR múltiple ofrece una configuración flexible de pequeños paneles en menos reacciones, por lo que conserva muestras valiosas. El multiplexado también aumenta el rendimiento del análisis, ya que necesita menos pocillos de la nanoplaca e incrementa el número de muestras que se pueden analizar en cada serie. Nuestros dPCR Microbial DNA Detection Assays pueden combinarse para los análisis múltiples.
Puede combinar sus propios ensayos para el multiplexado pidiendo ensayos individuales con colorantes distintos (FAM, HEX, TAMRA, ROX o Cy5). O bien, puede elegir entre nuestra oferta de paquetes de ensayos 5-plex, que nuestros científicos ya han probado en laboratorio experimental de dPCR. Otras combinaciones de los >700 ensayos prediseñados deben ser verificadas por su laboratorio.
Una comparación entre los análisis simple y múltiple con el uso de los dPCR Microbial DNA Detection Assays muestra una cuantificación similar y de gran precisión para todas las dianas (consulte la figura Detección microbiana en la dPCR múltiple en QIAcuity). Además de una cuantificación precisa de la diana, una serie dPCR 5-plex también muestra una detección altamente específica de diferentes patógenos microbianos del agua (véase la figura Detección de patógenos microbianos del agua en 5-plex en QIAcuity).
Principio
Los Microbial DNA Detection Assays prediseñados y los Custom dPCR Microbial Assays están destinados a utilizarse en la PCR digital de nanoplacas. Cada ensayo se basa en una amplificación de PCR de punto final de una región genética específica de la especie del microbio de interés o de una región de un gen microbiano individual. El producto amplificado se detecta con las sondas de hidrólisis fluorescentes específicas de la diana, lo que mejora la especificidad del ensayo.
Los dPCR Microbial DNA Detection Assays para la detección de especies bacterianas se dirigen a los genes del ARN ribosómico, sobre todo el gen de ARN ribosómico 16S, y se han diseñado con el uso de la base de datos GreenGenes para secuencias del 16S y las secuencias de ADN de la cepa tipo depositadas en el NCBI. Los ensayos para especies fúngicas, víricas y metazoarias tienen como objetivo diferentes regiones genéticas específicas, incluidos genes de ARN ribosómico y otros genes marcadores individuales, cada uno depositado en el NCBI. Se utilizaron diversas bases de datos para el diseño de ensayos para los genes de resistencia a antibióticos (p. ej., lahey.org, ARDB, etc.) y genes de factores de virulencia (p. ej., VFDB).
Los Custom dPCR Microbial Assays se crean con nuestro software de diseño de ensayos personalizado que se basa en un algoritmo sofisticado y probado exhaustivamente desarrollado específicamente para dianas microbianas. Garantiza que cada diseño de ensayo cumpla estrictos criterios de rendimiento para ofrecer ensayos robustos y de alta calidad con una sensibilidad y especificidad óptimas.
El principio de la reacción de dPCR en las nanoplacas se describe aquí.
Procedimiento
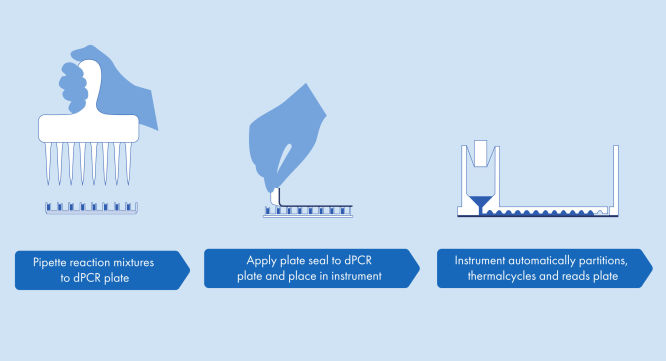
El protocolo del dPCR Microbial DNA Detection Assay y de los Custom dPCR Microbial Assays es sencillo y puede realizarse en cualquier laboratorio con el instrumento QIAcuity dPCR. Se aísla el ADN de la muestra y se añade a la QIAcuity Probe Mastermix lista para usar y al agua sin ADN microbiano (agua UCP). Esta mezcla se alicuota en cada pocillo de la preplaca de dPCR, que contiene juegos de cebadores y sondas de hidrólisis previamente dispensados. Las mezclas de reacción se transfieren desde la preplaca a los pocillos de una nanoplaca para dPCR, que después se sella y transfiere al instrumento QIAcuity dPCR (consulte la figura Flujo de trabajo simple y rápido basado en la placa). El instrumento QIAcuity dPCR realiza las funciones de división en partes, ciclado y obtención de imágenes de forma totalmente automatizada siguiendo los parámetros establecidos previamente. En función del protocolo de ciclado, los resultados de la serie de dPCR se pueden analizar en el QIAcuity Software Suite después de aproximadamente 2 horas (consulte la figura dPCR microbiana en aproximadamente 2 horas con tiempo mínimo de intervención manual).
Aplicaciones
Los dPCR Microbial DNA Detection Assays están especialmente pensados para la detección de especies bacterianas, fúngicas y víricas y de genes de resistencia a antibióticos microbianos o de factores de virulencia. Se puede analizar una variedad de muestras, incluidas heces, esputo, hisopo vaginal, aguas residuales y otras.
Además, la herramienta de diseño de Custom dPCR Microbial Assays permite diseñar a la perfección ensayos para cualquier diana microbiana (bacterias, hongos y virus) de interés que no esté cubierta por nuestros dPCR Microbial DNA Detection Assays prediseñados.
Paquetes de 5-plex probados en laboratorios experimentales de dPCR
| ID del paquete | Campo de aplicación | Números de catálogo de ensayos y fluoróforo* | Dianas (ID de la taxonomía del NCBI) |
| WW-001 | Agua residual 1 | DMA00278-F DMA00291-R DMA00340-H DMA00192-T DMA00344-C | Pseudomonas aeruginosa (287) Salmonella enterica (28901) Vibrio cholerae (666) Legionella pneumophila (446) Yersinia enterocolitica (630) |
| WW-002 | Agua residual 2 | DMA00340-H DMA00344-R DMA00199-C DMA00192-T DMA00710-F | Vibrio cholerae (666) Yersinia enterocolitica (630) Listeria monocytogenes (1639) Legionella pneumophila (446) Coronavirus humano SARS-CoV-2 (2697049) |
| WW-003 | Agua residual 3 | DMA00109-F DMA00192-H DMA00340-T DMA00194-R DMA00344-C | Clostridium perfringens (1502) Legionella pneumophila (446) Vibrio cholerae (666) Leptospira alexanderi (100053) Yersinia enterocolitica (630) |
| HM-001 | Microbioma humano 1 | DMA00148-F DMA00143-T DMA00024-R DMA00003-C DMA00150-H | Faecalibacterium prausnitzii (853) Eubacterium rectale (39491) Akkermansia muciniphila (239935) Acidaminococcus fermentans (951) Finegoldia magna (1260) |
| PB-001 | Probióticos 1 | DMA00177-F DMA00185-T DMA00061-C DMA00137-R DMA00320-H | Lactobacillus acidophilus (1579) Lactiplantibacillus plantarum (1590) Bifidobacterium bifidum (1681) Enterococcus faecium (1352) Streptococcus salivarius (1304) |
| RG-001 | Genes de resistencia 1 | DMA00566-F DMA00542-H DMA00576-T DMA00574-R DMA00575-C | Gen de resistencia a las fluoroquinolonas QnrS Grupo OXA-10 de betalactamasas de clase D Gen de la resistencia a la vancomicina vanB Gen de la bomba de expulsión de la tetraciclina tetA Gen de la bomba de expulsión de la tetraciclina tetB |
| RG-002 | Genes de resistencia 2 | DMA00557-T DMA00528-R DMA00548-F DMA00587-H DMA00553-C | Gen de resistencia a las fluoroquinolonas QepA Grupo de betalactamasas de clase B blaVIM-1 Grupo de betalactamasas de clase D OXA-48 Gen de resistencia a las sulfonamidas sul1 (43904) Grupo de betalactamasas de clase D OXA-58 |
| VG-001 | Genes de virulencia 1 | DMA00614-F DMA00635-T DMA00677-H DMA00680-R DMA00597-C | Subunidad fimbrial menor (fimH) Componente B de la hemolisina gamma (hlgB) Subunidad B de la toxina del tipo Shiga 1 codificada en el probacteriófago CP-933V Subunidad B de la toxina Shiga; subunidad de unión al receptor Enterotoxina accesoria del cólera (ace) |
| CP-001 | Producción de cánnabis 1 | DMA00278-F DMA00302-H DMA00365-R DMA00199-C | Pseudomonas aeruginosa (287) Staphylococcus aureus (1280) Aspergillus niger (5061) Listeria monocytogenes (1639) |
| HM-002 | Microbioma humano 2 | DMA00271-F DMA00017-T DMA00049-C DMA00142-H DMA00317-R | Prevotella oralis (28134) Actinomyces viscosus (1656) Bacteroides fragilis (817) Eubacterium infirmum (56774) Streptococcus oralis (1303) |
| RG-003 | Genes de resistencia 3 | DMA00579-F DMA00577-T DMA00573-R DMA00502-H DMA00559-C | aac(6')-Ib vanC oprm Grupo QnrB-1 Grupo CTX-M-1 |
| VG-002 | Genes de virulencia 2 | DMA00664-F DMA00677-H DMA00678-T DMA00642-R DMA00680-C | ply stx1B stx2A invA stxB |
| VG-003 | Genes de virulencia 3 | DMA00688-F DMA00668-H DMA00596-T DMA00597-R DMA00611-C | wbkA ptxA ace (E. faecalis) ace (V. cholerae) efaA |
* F: FAM, H: HEX, T: TAMRA, R: ROX o C: Cy5
Datos y cifras de respaldo
Flujo de trabajo simple y rápido basado en la placa
Las funciones de partición, termociclado y obtención de imágenes están todas integradas en un solo instrumento completamente automatizado que produce resultados en menos de dos horas. Los formatos de nanoplaca son susceptibles a la automatización inicial para la preparación de las muestras, lo que reduce aún más el tiempo de manipulación.