✓ 24/7 automatic processing of online orders
✓ Knowledgeable and professional Product & Technical Support
✓ Fast and reliable (re)-ordering
MinElute Reaction Cleanup Kit (50)
Cat. No. / ID: 28204
✓ 24/7 automatic processing of online orders
✓ Knowledgeable and professional Product & Technical Support
✓ Fast and reliable (re)-ordering
Features
- Very small elution volumes
- Fast procedure and easy handling
- High, reproducible recoveries
- Gel loading dye for convenient sample analysis
Product Details
The MinElute Reaction Cleanup Kit provides spin columns, buffers, and collection tubes for silica membrane-based purification of DNA 70 bp – 4 kb in size from enzymatic reactions. The spin columns are designed to allow elution in very small volumes (as little as 10 µl), delivering highly concentrated DNA in high yields. An integrated pH indicator allows easy determination of the optimal pH for DNA binding to the spin column. The procedure can be fully automated on the QIAcube Connect. DNA fragments purified with the MinElute system are ready for direct use in all applications, including sequencing, microarray analysis, ligation and transformation, restriction digestion, labeling, microinjection, PCR, and in vitro transcription.
For optimal results it is recommended to use this product together with QIAvac 24 Plus.
Performance
The MinElute Reaction Cleanup Kit ensures cleanup of up to 5 µg DNA (70 bp to 4 kb) from enzymatic reactions, delivering high yields of DNA suitable for a range of applications. The kit provides spin columns for cleanup of enzymatic reactions. Using a microcentrifuge or vacuum manifold, high concentration of DNA fragment (70 bp – 4 kb) is quickly achieved. (DNA fragments larger than 4 kb should be purified using the QIAquick System.)
| Protein | Molecular weight per enzyme subunit (kDa) |
|---|---|
| DNA Polymerase I | 109 |
| Klenow fragment | 62 |
| Calf intestinal alkaline phosphatase | 69 |
| T4 DNA ligase | 55 |
| T4 Polynucleotide kinase | 35 |
| Terminal transferase | 32 |
| DNase I | 31 |
| Restriction enzymes | Varies |
Principle
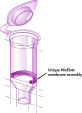
MinElute Kits contain a silica membrane assembly for binding of DNA in high-salt buffer and elution with low-salt buffer or water. The purification procedure removes primers, nucleotides, enzymes, mineral oil, salts, agarose, ethidium bromide, and other impurities from DNA samples. Silica-membrane technology eliminates the problems and inconvenience associated with loose resins and slurries. Specialized binding buffers are optimized for specific applications and promote selective adsorption of DNA molecules within particular size ranges.
Gel loading dye
To enable faster and more convenient sample processing and analysis, gel loading dye is provided. GelPilot Loading Dye contains three tracking dyes (xylene cyanol, bromophenol blue, and orange G) to facilitate the optimization of agarose gel run time and prevent smaller DNA fragments migrating too far (see figure " GelPilot Loading Dye").
See figures
Procedure
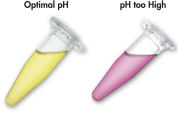
The MinElute system uses a simple bind-wash-elute procedure (see flowchart " MinElute procedure"). Binding buffer is added directly to the enzymatic reaction, and the mixture is applied to the MinElute spin column. The binding buffer contains a pH indicator, allowing easy determination of the optimal pH for DNA binding (see figure "pH Indicator Dye"). Nucleic acids adsorb to the silica membrane in the high-salt conditions provided by the buffer. Impurities are washed away and pure DNA is eluted with a small volume of low-salt buffer provided or water, ready to use in subsequent applications.
Handling
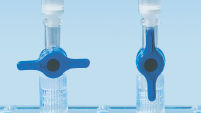
MinElute spin columns are designed to provide two convenient handling options (see flowchart "MinElute procedure"). The spin columns fit into a conventional table-top microcentrifuge or onto any vacuum manifold with luer connectors, such as QIAvac 24 Plus or QIAvac 6S with QIAvac Luer Adapters. The MinElute Reaction Cleanup Kit, in addition to other QIAGEN spin-column-based kits, can be fully automated on the QIAcube Connect, enabling increased productivity and standardization of results (see figures "Spin column handling options A, B, C, D, and E" and " QIAcube Connect").
See figures
Applications
DNA fragments purified with the MinElute System are ready for direct use in all applications, including:
- Sequencing
- Microarray analysis
- Ligation and transformation
- Restriction digestion
- Labeling
Supporting data and figures
Spin column handling options — E.

Specifications
| Features | Specifications |
|---|---|
| Binding capacity | 5 µg |
| Fragment size | 70 bp – 4 kb |
| Processing | Manual |
| Elution volume | 10 µl |
| Recovery: oligonucleotides dsDNA | Recovery: oligonucleotides, dsDNA |
| Removal <10mers 17–40mers dye terminator proteins | Removal <40mers |
| Format | Tube |
| Sample type: applications | DNA, oligonucleotides: Enzymatic reactions |
| Technology | Silica technology |