✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
QIAEX II Gel Extraction Kit (150)
Cat. No. / ID: 20021
✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
Eigenschaften
- Effiziente Extraktion von DNA von 40 bp bis 50 kb
- Gelextraktion aus TAE- oder TBE-Agarose- und Polyacrylamidgelen
- Enthält kein Natriumjodid, das nachfolgende Reaktionen stören könnte
- Keine Scherung großer DNA-Fragmente
Angaben zum Produkt
Das QIAEX II System enthält eine Silikapartikel-Suspension, an die sich DNA-Fragmente in Gegenwart chaotroper Salze binden. Die QIAEX II Suspension wird Lösungen oder aufgelösten Agarosegelschnitten zugesetzt und bindet DNA. Die Partikel werden durch einen kurzen Zentrifugationsschritt gewonnen und gewaschen. DNA von 40 bp bis 50 kb wird in Tris-Puffer oder Wasser eluiert.
Leistung
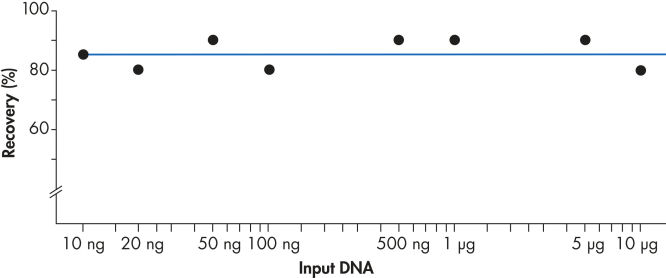
Mit dem QIAEX II System werden 10 ng bis 10 µg DNA effizient zurückgewonnen (siehe Abbildung „Konsistente Rückgewinnung“). Das vielseitige Verfahren für die Batch-Aufreinigung von Gelfragmenten lässt sich mit 30 µl QIAEX II Suspension leicht auf eine Bindekapazität von 15 µg hochskalieren.
Das QIAEX II System enthält Silika-Partikel für die Aufreinigung von 60–95 % DNA-Fragmenten (40 bp – 50 kb). Ein Volumen von 10 µl QIAEX II Suspension bindet bis zu 5 µg DNA, die anschließend in 20 µl eluiert wird.
Rückgewinnung nach Größe
| DNA-Größe | Rückgewinnung, Prozent* |
|---|---|
| 44 bp | 75 |
| 75 bp | 75 |
| 500 bp | 95 |
| 7,5 kb | 85 |
| 23,5 kb | 75 |
| 48,5 kb | 60 |
Abbildungen ansehen
Prinzip
Die Aufreinigung von DNA-Fragmenten mit dem QIAEX II System basiert auf der Solubilisierung von Agarose und der selektiven Adsorption von Nukleinsäuren an QIAEX II-Silikagelpartikel in Gegenwart von chaotropem Salz. QIAEX II trennt DNA von Salzen, Agarose, Polyacrylamid, Farbstoffen, Proteinen und Nukleotiden ohne Phenolextraktion oder Ethanolpräzipitation. QIAEX II eignet sich für jede Art von Agarose in TAE- oder TBE-Puffern.
QIAEX II-Partikel bilden eine Suspension für die Gelextraktion und gewährleisten die effiziente Rückgewinnung ohne Scherung, selbst bei großen DNA-Fragmenten. Optimierte Puffer ermöglichen die DNA-Rückgewinnung ohne Natriumjodid, das sich nur schwer aus DNA-Proben entfernen lässt und nachfolgende Reaktionen beeinträchtigen kann.
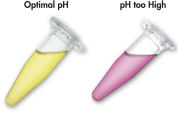
Der mit dem QIAEX II System verwendete Solubilisierungs- und Bindepuffer enthält einen einzigartigen pH-Indikator. Ein einfacher Farbwechsel zeigt an, ob der pH-Wert der Bindungsmischung für eine effiziente Adsorption der DNA an die QIAEX II-Silikapartikel optimal ist (siehe Abbildung „pH-Indikatorfarbstoff“). Der Farbstoff ermöglicht auch die einfache Visualisierung nicht aufgelöster Agarose in der Bindungsmischung und gewährleistet so die vollständige Solubilisierung und damit eine maximale Ausbeute.
Der pH-Indikatorfarbstoff im Lösungs- und Bindepuffer erlaubt die einfache visuelle Bestimmung des optimalen pH-Werts für die DNA-Adsorption (pH ≤7,5). Bei häufig verwendetem oder falsch angesetztem Agarosegel-Elektrophoresepuffer kann das Bindungsgemisch einen falschen pH-Wert aufweisen. In diesem Fall lässt sich der pH-Wert leicht durch Zugabe von 10 µl 3 M Natriumacetat, pH 5,0, einstellen.
Abbildungen ansehen
Verfahren
QIAEX II-Silikagelpartikel werden zum aufgelösten Gelschnitt hinzugefügt, und die Partikel werden durch einen kurzen Zentrifugationsschritt gewonnen (siehe Flussdiagramm „QIAEX II-Verfahren“). Nach dem Waschen wird das aufgereinigte DNA-Fragment in 20 µl Tris-Puffer oder Wasser eluiert.
Das QIAEX II System enthält die QIAEX II-Suspension zusammen mit Binde- und Waschpuffern und einem umfassenden Handbuch. Es enthält Protokolle für die Aufreinigung von DNA aus Agarosegelen, Lösungen und Polyacrylamidgelen.
Abbildungen ansehen
Anwendungen
Die mit dem QIAEX II System aufgereinigte DNA lässt sich direkt für die meisten Anwendungen verwenden, darunter:
- Restriktionsverdau
- Markierung
- Ligation
- PCR
| Eigenschaften | Spezifikationen |
|---|---|
| Bindekapazität | 5 µg/10 µl |
| Elutionsvolumen | 20 µl |
| Format | Röhrchen |
| Fragmentgröße | 40 bp – 50 kb |
| Verfahren | Manuell |
| Rückgewinnung: Oligonukleotide, dsDNA | Rückgewinnung: dsDNA-Fragmente |
| Entfernung von < 10mer- und 17- bis 40mer-Farbstoffterminator-Proteinen | Entfernung von < 40meren |
| Probentyp: Applikationen | DNA: PCR-Reaktionen |
| Technologie | Silika-Technologie |
Ergänzende Daten und Abbildungen
Konsistente Rückgewinnung