✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
E. coli resDNA Quant Standard Kit
Cat. No. / ID: 250221
✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
Eigenschaften
- Vorgemischter Master-Mix mit Kontrollen für einfachen Reaktionsaufbau und Nachweis von Wirtszell-DNA
- Ermöglicht den genauen Nachweis von Wirtszell-DNA-Rückständen aus E.coli-, CHO- und HEK293-Zellen bis zu niedrigen Femtogramm-Mengen
- Ein gezielter Multi-copy-Assay, der stark fragmentierte Wirtszell-DNA nachweist und quantifiziert
- Ermöglicht den Nachweis von Wirtszell-DNA in extrahierten und nicht extrahierten Proben
Angaben zum Produkt
Die Kontamination mit Wirtszell-DNA in Produkten wie Impfstoffen, Medikamenten und anderen Biopharmazeutika birgt erhebliche Gesundheitsrisiken. Darum unterliegen sichere Grenzwerte einer strengen Regulierung durch Behörden wie die US FDA und die WHO. Klare Richtlinien für die Obergrenzen von Rest-DNA werden basierend auf dem Verabreichungsweg von Medikamenten, der Infektiösität und der Onkogenität der kontaminierenden Zell-DNA festgelegt. Beispielsweise sollte die parenterale Gabe nicht tumorigener Zell-DNA auf 10 ng/Dosis und eine maximale Länge von 200 bp begrenzt werden, während die WHO für die orale Gabe von Impfstoffen weniger als 100 µg Rest-DNA pro Dosis empfiehlt.
Nachweis und Entfernung derartiger Kontaminationen bei der Arzneimittelherstellung erfordern hochsensitive und genaue Messungen der extrem geringen Mengen spezifischer Wirtszell-DNA in den Arzneimitteln.
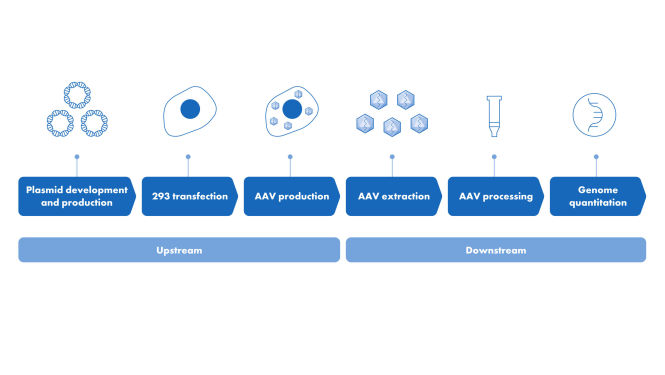
Digitale PCR ist die bevorzugte Nachweismethode zur Quantifizierung von Rest-DNA. Grund dafür sind ihre beispiellose Sensitivität und Genauigkeit, insbesondere bei Kontaminationen im Spurenbereich, verglichen mit anderen Nachweismethoden wie qPCR. QIAcuity Residual DNA Quantification Kits ermöglichen den genauen und präzisen Nachweis von Wirtszell-DNA aus Escherichia coli (E. coli), Chinese Hamster Ovary(CHO)-Zellen und Human Embryonic Kidney 293(HEK293)-Zellen.
Leistung
Die QIAcuity Residual DNA Quantification Kits liefern genaue Quantifizierungsergebnisse für CHO-, E. coli- und HEK293-Rest-DNA (resDNA) mit oder ohne Extraktion, selbst in Gegenwart von PCR-Kontaminationen oder inhibitorischen Reagenzien. Bei den Assays handelt es sich um gezielte Multi-copy-Assays, die stark fragmentierte Wirtszell-DNA-Rückstände genau bestimmen.
| Assay | Target-Kopienzahl | Amplifikatgröße | Umrechnungsfaktor Kopien/µl in fg/µl |
|---|---|---|---|
| QIAcuity E.coli resDNA Quant Kit | 7 | < 200 | 0,35 |
| QIAcuity CHO resDNA Quant Kit | ~ 1 Million, nicht definiert (wiederholtes Element) | < 100 | 0,28 |
| QIAcuity HEK293 resDNA Quant Kit | ~ 1 Million, nicht definiert (wiederholtes Element) | < 100 | 1,54 |
Das QIAcuity E.coli resDNA Quant Kit weist geringe Mengen ab 5 fg Rest-DNA in einer Einzel-Reaktion nach
| Beladungsmenge je Reaktion (fg/rxn) | E.coli-Standard (Kopien/µl) | Interne Kontrolle (Kopien/µl)* |
|---|---|---|
| 50000 | 2949,7 | 99,9 |
| 5000 | 291,6 | 91,7 |
| 500 | 27,6 | 91,4 |
| 50 | 2,8 | 93,2 |
| 25 | 1,46 | 92,5 |
| 5 | 0,45 | 90,6 |
| NTC | 0 | 98,9 |
*100 Kopien/µl für interne Kontrolle erwartet
Die digitale PCR bietet im Vergleich zur qPCR eine höhere Nachweissensitivität bei niedrigeren Template-Einsatzmengen und ermöglicht damit eine robustere Anwendung.
Das QIAcuity CHO resDNA Quant Kit weist geringe Mengen ab 5 fg Rest-DNA in einer Einzel-Reaktion nach
| Beladungsmenge je Reaktion (fg/rxn) | CHO-Standard (Kopien/µl) | Interne Kontrolle (Kopien/µl)* |
|---|---|---|
| 50000 | 4939,3 | 108,7 |
| 5000 | 508,3 | 104,9 |
| 500 | 50,2 | 97,6 |
| 50 | 4,7 | 99,5 |
| 25 | 2,6 | 101,3 |
| 5 | 0,6 | 100,4 |
| NTC | 0 | 104,7 |
*100 Kopien/µl für interne Kontrolle erwartet
Das QIAcuity HEK293 resDNA Quant Kit weist geringe Mengen ab 5 fg Rest-DNA in einer Einzel-Reaktion nach
| Beladungsmenge je Reaktion (fg/rxn) | HEK293-Standard (Kopien/µl) | Interne Kontrolle (Kopien/µl)* |
|---|---|---|
| 50000 | 1104,9 | 96,6 |
| 5000 | 104,5 | 92,5 |
| 500 | 8,83 | 92,4 |
| 50 | 0,99 | 94,4 |
| NTC | 0 | 96,2 |
*100 Kopien/µl für interne Kontrolle erwartet
Die Kits werden zusammen mit dem QIAcuity Digital PCR System und QIAcuity Nanoplates verwendet und ermöglichen einen lückenlosen, schnellen dPCR-Workflow, der mit der qPCR vergleichbar ist, aber eine absolute Quantifizierung der resDNA in Ihrer Probe ermöglicht. Die Kits wurden unter Berücksichtigung der Anforderungen an Bioprozessherstellung und -QK entwickelt.
Prinzip
Das Prinzip der dPCR-Reaktion in den Nanoplatten finden Sie hier beschrieben.
Die Verwendung der digitalen PCR mit QIAcuity zur Überwachung von Wirtszell-DNA verbessert den LOD/LOQ-Wert durch Partitionierung der Bulk-Probe mithilfe der QIAcuity Nanoplate 26k. Die Partitionierung steigert die effektive Konzentration des Targets, sodass eine geringere Menge an Wirtszell-DNA erfasst und mit höherer Genauigkeit und Präzision gemessen werden kann. Die Möglichkeit, hohe Template-Einsatzmengen in die QIAcuity Nanoplate 26k zu laden, in Kombination mit einer erhöhten Anzahl an Partitionen, ist einer der Gründe für die höhere Genauigkeit und Präzision des Nachweises von Wirtszell-Resten.
Verfahren
Das Lysat wird dem Rest-Quantifizierungsassay zusammen mit der internen Kontrolle zugegeben und die Wirtszell-DNA wird mittels absoluter Quantifizierung gemessen. Kopien/µl werden über einen bereitgestellten Umrechnungsfaktor in fg/µl umgerechnet.
Anwendungen
Die QIAcuity Residual DNA Quantification Kits eignen sich ideal zur hochpräzisen Quantifizierung von Wirtszell-DNA in komplexen Zwischenprodukten von Bioverfahren.
Ergänzende Daten und Abbildungen
In der finalen Substanz sind nur begrenzte Mengen an Verunreinigungen erlaubt
Die Verschleppung von Wirtszell-DNA stellt ein Sicherheitsrisiko dar und unterliegt einer strengen Regulierung durch Behörden wie FDA und WHO