✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
MinElute PCR Purification Kit (50)
Cat. No. / ID: 28004
✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
Eigenschaften
- Sehr kleine Elutionsvolumen
- Schnelles Verfahren und einfache Handhabung
- Hohe, reproduzierbare Rückgewinnung
- Gel-Ladefarbstoff für komfortable Probenanalyse
Angaben zum Produkt
Das MinElute PCR Purification Kit enthält Spin-Säulen, Puffer und Sammelröhrchen für die Silikamembran-basierte Aufreinigung von PCR-Produkten mit einer Größe von 70 bp bis 4 kb. Die Spin-Säulen wurden für die Elution in sehr kleinen Volumen (bis zu 10 μl) entwickelt und liefern hohe Ausbeuten an hochkonzentrierter DNA. Ein optionaler pH-Indikator ermöglicht die einfache Bestimmung des optimalen pH-Werts für die DNA-Bindung an die Spin-Säule. Das Verfahren kann auf dem QIAcube Connect vollständig automatisiert werden.
Leistung
Das MinElute PCR Purification Kit enthält Spin-Säulen für das Cleanup von PCR-Produkten. Mithilfe einer Mikrozentrifuge oder eines Vakuumverteilers können schnell hohe Konzentrationen an DNA-Fragmenten (70 bp – 4 kb) erreicht werden. (DNA-Fragmente größer als 4 kb sollten mit dem QIAquick PCR Purification Kit aufgereinigt werden.)
Abbildungen ansehen
Prinzip
Das MinElute PCR Purification Kit enthält eine Silika-Membraneinheit für die Bindung von DNA in Hochsalzpuffer und die Elution mit Niedrigsalzpuffer oder Wasser. Die Silika-Membrantechnologie beseitigt die Probleme und Schwierigkeiten, die mit losen Harzen und Suspensionen verbunden sind.
Gel-Ladefarbstoff
Der mitgelieferte Gel-Ladefarbstoff ermöglicht eine schnellere und bequemere Verarbeitung und Analyse der Proben. Der GelPilot Loading Dye enthält drei Farbstoffe zur Nachverfolgung (Xylencyanol, Bromphenolblau und Orange G), um die Optimierung der Agarosegel-Laufzeit zu erleichtern und zu verhindern, dass kleinere DNA-Fragmente zu weit migrieren (siehe Abbildung „ GelPilot Loading Dye“).
Abbildungen ansehen
Verfahren
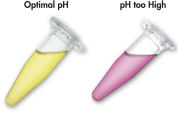
Das MinElute System nutzt ein einfaches Verfahren mit den Schritten „Binden-Waschen-Eluieren“. Der Bindepuffer wird direkt zur PCR-Probe oder einer anderen enzymatischen Reaktion hinzugefügt, und die Mischung wird auf die MinElute Spin Column aufgetragen. Zur einfachen Bestimmung des optimalen pH-Werts für die DNA-Bindung enthält der Bindepuffer einen pH-Indikator (siehe Abbildung „pH-Indikatorfarbstoff“). Die Nukleinsäuren werden unter den Hochsalzbedingungen des Puffers an die Silikamembran adsorbiert. Verunreinigungen werden durch Waschen entfernt, und die reine DNA wird in einem kleinen Volumen des mitgelieferten Niedrigsalzpuffers oder mit Wasser eluiert und kann direkt für nachfolgende Anwendungen verwendet werden.
Handhabung
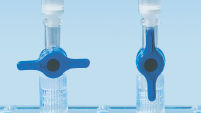
MinElute Spin Columns sind so konzipiert, dass sie zwei praktische Handhabungsoptionen bieten (siehe Flussdiagramm „MinElute-Verfahren“). Die Spin-Säulen passen in eine herkömmliche Tisch-Mikrozentrifuge oder auf jeden Vakuumverteiler mit Luer-Anschlüssen, beispielsweise QIAvac 24 Plus, und können außerdem auf dem QIAcube Connect vollständig automatisiert werden (siehe Abbildungen „Handhabungsoptionen für Spin-Säulen A und B“ und „ QIAcube Connect“).
Abbildungen ansehen
Anwendungen
Die mit dem MinElute System aufgereinigten DNA-Fragmente lassen sich direkt für alle Anwendungen verwenden, darunter:
- Sequenzierung, einschließlich Next-Generation Sequencing
- Microarray-Analyse
- Ligation und Transformation
- Restriktionsverdau
- Markierung
Ergänzende Daten und Abbildungen
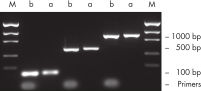
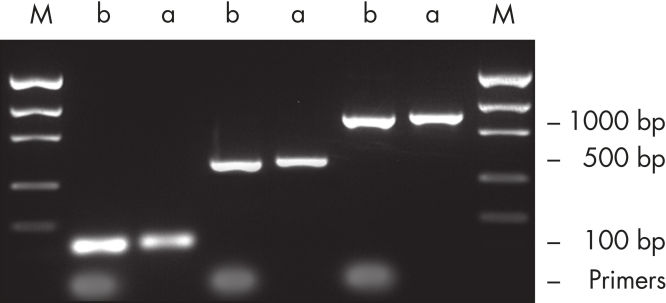
Effiziente Primerentfernung
Specifications
| Features | Specifications |
|---|---|
| Binding capacity | 5 µg |
| Sample type: applications | DNA, Oligonukleotide: PCR-Reaktionen |
| Elution volume | 10 µl |
| Fragment size | 70 bp – 4 kb |
| Recovery: oligonucleotides dsDNA | Rückgewinnung: Oligonukleotide, dsDNA |
| Format | Röhrchen |
| Technology | Silika-Technologie |
| Processing | Manual |
| Removal <10mers 17–40mers dye terminator proteins | Entfernung von < 40meren |