✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
QIAquick 96 PCR Purification Kit (4)
Cat. No. / ID: 28181
✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
Eigenschaften
- Rückgewinnung von gebrauchsfertiger DNA von bis zu 95 %
- Schnelles und praktisches Verfahren
- Reinigung von DNA von bis zu 10 kb Länge in drei einfachen Schritten
Angaben zum Produkt
QIAquick 96 PCR Purification Kits enthalten 96-Well-Platten, Puffer und Sammelröhrchen für die Silikamembran-basierte Hochdurchsatzaufreinigung von PCR-Produkten mit einer Größe > 100 bp. DNA mit einer Größe von bis zu 10 kb wird in einem einfachen und schnellen Verfahren („Binden-Waschen-Eluieren“) aufgereinigt und in einem Volumen von 60–80 µl (resultierend in einem Elutionsvolumen von 40–60 µl) eluiert. Das Reinigungsverfahren kann auf der BioRobot Universal Workstation mit dem QIAquick 96 PCR BioRobot Kit vollständig automatisiert werden.
Leistung
Abbildungen ansehen
Prinzip
QIAquick 96 Kits enthalten eine Silika-Membraneinheit für die Bindung von DNA in Hochsalzpuffer und die Elution mit Niedrigsalzpuffer oder Wasser. Durch die Aufreinigung werden Primer, Nukleotide, Enzyme, Mineralöl, Salze, Agarose, Ethidiumbromid und andere Verunreinigungen aus DNA-Proben entfernt. Die Silika-Membrantechnologie beseitigt die Probleme und Schwierigkeiten, die mit losen Harzen und Suspensionen verbunden sind. Spezialisierte Bindepuffer sind für spezifische Anwendungen optimiert und fördern die selektive Adsorption von DNA-Molekülen innerhalb bestimmter Größenbereiche.
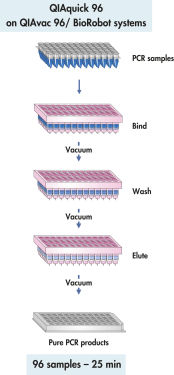
Das QIAquick 96-Verfahren ermöglicht die parallele Aufreinigung von bis zu 96 PCR-Proben mittels effizienter vakuumgestützter Aufreinigung auf einem QIAvac 96.
Das QIAquick 96 PCR BioRobot Kit ist ein speziell für den Einsatz auf dem BioRobot Universal optimiertes Kitformat. Das Kit enthält QIAquick 96 Module sowie alle Puffer und Plastikmaterialien, die für die automatisierte Hochdurchsatzreinigung von 96 PCR-Proben benötigt werden.
Verfahren
Das QIAquick System nutzt ein einfaches Verfahren mit den Schritten „Binden-Waschen-Eluieren“ (siehe Flussdiagramm „ QIAquick 96-Verfahren“). Der Bindepuffer wird der PCR-Probe oder einer anderen enzymatischen Reaktion direkt zugesetzt und die Mischung wird anschließend auf die 96-Well-Platte aufgetragen. Die Nukleinsäuren werden unter den Hochsalzbedingungen des Puffers an die Silikamembran adsorbiert. Verunreinigungen werden durch Waschen entfernt, und die reine DNA wird in einem kleinen Volumen des mitgelieferten Niedrigsalzpuffers oder mit Wasser eluiert und kann direkt für nachfolgende Anwendungen verwendet werden.
Handhabung
QIAquick Multiwell Module werden mittels vakuumgestützter Aufreinigung auf QIAvac Verteilern verarbeitet. Das QIAquick 96 PCR Purification Kit erfordert den Einsatz des QIAvac 96-Vakuumverteilers. Das Reinigungsverfahren kann auf den BioRobot U Workstations mit dem QIAquick 96 PCR BioRobot Kit vollständig automatisiert werden.
Abbildungen ansehen
Anwendungen
Die mit dem MinElute oder QIAquick System aufgereinigten DNA-Fragmente können direkt für zahlreiche Anwendungen genutzt werden, darunter Sequenzierung, Microarray-Analyse, Ligation und Transformation, Restriktionsverdau, Markierung, Mikroinjektion, PCR und In-vitro-Transkription.
Ergänzende Daten und Abbildungen
QIAquick 96-Verfahren
Das QIAquick 96 Kit nutzt ein Verfahren mit den Schritten „Binden-Waschen-Eluieren“ auf der QIAvac 96 oder dem BioRobot Universal System.
Specifications
| Features | Specifications |
|---|---|
| Binding capacity | 10 µg |
| Processing | Manuell/automatisiert |
| Removal <10mers 17–40mers dye terminator proteins | Entfernung von < 40meren |
| Sample type: applications | DNA, Oligonukleotide: PCR-Reaktionen |
| Format | 96-Well-Platte |
| Fragment size | 100 bp – 10 kb |
| Technology | Silika-Technologie |
| Recovery: oligonucleotides dsDNA | Rückgewinnung: Oligonukleotide, dsDNA |
| Elution volume | 60–80 µl |