EpiTect Hi-C Kit
For high-resolution mapping of chromatin folding, high-quality assembly of genome sequences, haplotype phasing and identifying chromosomal rearrangements
For high-resolution mapping of chromatin folding, high-quality assembly of genome sequences, haplotype phasing and identifying chromosomal rearrangements
✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
Cat. No. / ID: 59971
✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
Hi-C wurde ursprünglich als leistungsstarke Technik zur genomweiten Erfassung der Chromosomenkonformation entwickelt, die die Charakterisierung der Chromatinfaltung mit kb-Auflösung ermöglicht. Die Technik hat aber auch andere wichtige Anwendungen. So wird Hi-C beispielsweise zur Erstellung von hochgradig kontingenten Genomassemblierungen mit wenigen und sehr langen Gerüsten aus Organismen ohne bekanntes Referenzgenom verwendet. Darüber hinaus ist Hi-C auch sehr nützlich für die Haplotyp-Phasierung und den Nachweis von chromosomalen Rearrangements.
Das EpiTect Hi-C Kit beruht auf einem robusten, aber dennoch einfachen und schnellen Protokoll mit geringen Anforderungen an den Zelleinsatz. Es ermöglicht die Generierung von hochwertigen Hi-C Illumina NGS-Bibliotheken aus vernetzten Zellen in weniger als 2 Tagen.
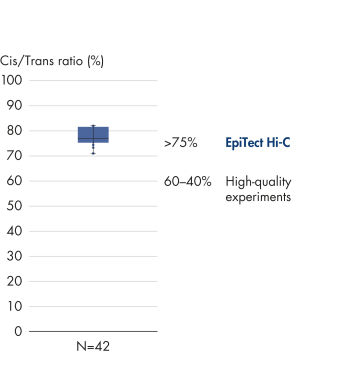
Das EpiTect Hi-C Kit dient zur Erstellung qualitativ hochwertiger Hi-C-NGS-Bibliotheken und stellt damit sicher, dass aus der kostspieligen nachgelagerten Tiefensequenzierung erstklassige Daten gewonnen werden. Die Sequenzierergebnisse aus mehr als 40 EpiTect Hi-C-Bibliotheken wurden analysiert, um die Leistung des Kits zu bewerten. Die wichtigsten QK-Kennzahlen sind in den folgenden Abbildungen dargestellt: Prozentsatz der Hi-C-Ereignisse, Prozentsatz der langreichweitigen cis-Interaktionen, cis/trans-Verhältnis, Kein Bias der Strangorientierung mit dem EpiTect Hi-C Kit und Prozentsatz der gepaarten Reads, die von einem einzigen Restriktionsfragment stammen. Die mit dem EpiTect Hi-C Kit gewonnenen Daten zeigen, dass die mit dem Kit erzeugten NGS-Bibliotheken im Durchschnitt weit über die Kriterien hinausgehen, die normalerweise für ein erfolgreiches Hi-C-Experiment als ausreichend gelten.
Beim Hi-C handelt es sich um einen Proximity-Ligation-Assay, der Chromatin-Interaktionen für das gesamte Genom erfasst. Das EpiTect Hi-C Kit ist eine spezielle DNA-Präparationsmethode, die die Erstellung einer Illumina-kompatiblen NGS-Bibliothek ermöglicht (siehe Abbildungen EpiTect Hi-C-Workflow – Tag 1 und EpiTect Hi-C-Workflow – Tag 2). Kurz beschrieben beginnt der Assay mit der Aufreinigung der Kerne, in denen die Chromatinkonformation durch die chemische Vernetzung der DNA-Bindungsproteine und der DNA eingefroren wurde. Die DNA wird dann mit einem 4-bp-Restriktionsenzym vollständig verdaut. Offene DNA-Enden werden mit Biotin markiert und anschließend ligiert. Die Paired-End-Sequenzierung der Hi-C-Ligationsprodukte ermöglicht die Detektion einer sehr großen Anzahl chimärer Sequenzen aus DNA-Strängen, die räumlich eng miteinander verbunden waren. Die Wahrscheinlichkeit, dass zwei Sequenzen miteinander ligiert werden, hängt von ihrem mittleren räumlichen Abstand ab. Die Quantifizierung von Ligationsverbindungen ermöglicht die Bestimmung von DNA-Kontakthäufigkeiten, anhand derer eine hochauflösende Kartierung der Chromatinfaltung erfolgen kann.
Der EpiTect Hi-C-Workflow (siehe Abbildungen EpiTect Hi-C-Workflow – Tag 1 und EpiTect Hi-C-Workflow – Tag 2) stellt eine deutliche Verbesserung gegenüber bisher veröffentlichten Protokollen dar. Ein wochenlanges und kompliziertes Verfahren wurde in ein einfaches und robustes Protokoll umgewandelt, das in nur 1,5 Tagen durchgeführt werden kann. Darüber hinaus wurde die erforderliche Probenmenge um eine Größenordnung reduziert, sodass Hi-C-NGS-Bibliotheken aus nur 5000 Zellen erstellt werden können. Das Protokoll wurde für die Arbeit mit vernetzten Zellen aus Säugetierzellkulturen entwickelt.
Das EpiTect Hi-C-Verfahren ist eine Version der in situ (d. h. im Zellkern) durchgeführten Hi-C-Methode, bei der die Kerne schonend aufgereinigt und permeabilisiert werden, um den räumlichen Aufbau des Genoms während der ersten Verdauungs- und Ligationsschritte zu erhalten. Dieser Prozess ist von entscheidender Bedeutung, um das Hintergrundrauschen durch nicht-aussagekräftige Ligationsereignisse zu minimieren, die für den Aufbau des Genoms nicht relevant sind. Dies liegt daran, dass intakte Zellkerne die Bewegung und zufälligen Kollisionen von vernetzten Komplexen einschränken, sodass Ligationsereignisse vorwiegend zwischen topologisch assoziierten DNA-Fragmenten stattfinden.
Erstellung von Hi-C-NGS-Bibliotheken
Der Workflow des EpiTect Hi-C Kits besteht aus 2 Teilen, die jeweils an einem Tag durchgeführt werden können. Die Schritte des Protokolls sind in den folgenden Tabellen zusammengefasst und in den Abbildungen EpiTect Hi-C-Workflow – Tag 1 und EpiTect Hi-C-Workflow – Tag 2 grafisch dargestellt. Die im Lieferumfang enthaltenen Illumina-Adapter haben Sequenz-Barcodes, die die Multiplex-Sequenzierung von bis zu 6 Proben ermöglichen.
Das vollständige Protokoll finden Sie in unserem ausführlichen EpiTect-Hi-C-Handbuch.
Datenanalyse
Die Hi-C-Datenanalyse wird in unserem GeneGlobe Data Analysis Center angeboten. Die Ergebnisse der Hi-C-Sequenzierung können mithilfe des EpiTect Hi-C Data Analysis Portals analysiert werden, das unter Verwendung von Open-Source-Tools einen QK-Sequenzierungsbericht, Hi-C-Kontaktmatrizen und eine grafische Darstellung von Chromatinkontaktkarten bereitstellt. Weitere Informationen finden Sie in unserer Bedienungsanleitung für das EpiTect Hi-C Data Analysis Portal.
Chromatinkonformation
Hi-C hat sich in kürzester Zeit zu einem sehr wichtigen Instrument für die Analyse des nukleären Aufbaus entwickelt. Die Analyse der Hi-C-Daten hat die erstaunliche Komplexität der Genomstruktur offenbart, mit mehreren Ebenen der räumlichen Organisation, die das Genom mit zunehmender Auflösung in Chromosomenterritorien, chromosomale Unterkompartimente, topologisch assoziierte Domänen (TADs) und DNA-Schleifen aufteilen (siehe Abbildung Ebenen des Chromatinaufbaus). Darüber hinaus ist der Aufbau des Genoms dynamisch und verändert sich im Laufe der Entwicklung. Es gibt keine Zelltypen, bei denen die Chromosomen gleich gefaltet sind.
Chromosomale Rearrangements und Kopienzahlvarianten
Einzelne Chromosomen sind physisch in diskrete Territorien aufgeteilt. Daher finden die von Hi-C erfassten DNA-Interaktionen in erster Linie zwischen der DNA desselben Chromosoms (in cis) und nur in geringem Maße zwischen den Chromosomen (in trans) statt. Aufgrund dieses Phänomens kann Hi-C als Assay für das gesamte Genom verwendet werden, um Translokationen und andere strukturelle Varianten von Interesse zu identifizieren. Im Vergleich zu anderen NGS-Techniken erfordert Hi-C eine extrem niedrige Abdeckung, wodurch Kosten gespart werden können. Außerdem können Rearrangements, die schlecht kartierbare Regionen betreffen, mit Hi-C besser detektiert werden als mit NGS-Standardmethoden. Praktisch ist, dass dieselben Hi-C-Daten auch zum Nachweis von Kopienzahländerungen verwendet werden können.
Genomassemblierung – Haplotyp-Phasierung
Bei der Sequenzierung und Assemblierung der Genome neuer Arten wird die Erstellung von Sequenzgerüsten oft durch große Abschnitte mit sich wiederholenden Sequenzen eingeschränkt, die über die Reichweite der Sequenzierung hinausgehen. In den Hi-C-Daten findet die überwiegende Mehrheit der Interaktionen in cis zwischen Loci auf demselben Chromosom statt. Außerdem findet ein erheblicher Teil dieser cis-Interaktionen über große Entfernungen statt, nämlich zwischen Loci, die durch Millionen von DNA-Basen voneinander getrennt sind. Diese Eigenschaften von Chromatin-Interaktionen können genutzt werden, um Sequenzgerüste zu ordnen, zu orientieren und zu nahezu vollständigen Chromosomen zusammenzufügen, ohne dass dafür ein Referenzgenom erforderlich ist. Nach dem gleichen Prinzip können Hi-C-Interaktionskarten zur Erstellung diploider Genome verwendet werden, indem genetische Varianten den väterlichen und mütterlichen Schwesterchromosomen zugeordnet werden (siehe Abbildung Nachgelagerte Anwendungen von Hi-C-Sequenzierungsdaten).