✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
dPCR Microbial DNA Detection Assays
Cat. No. / ID: 250207
✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
Eigenschaften
- Vorgefertigte Assays für den Nachweis von mikrobiellen Spezies, Virulenzgenen oder Antibiotikaresistenzgenen
- Assays für mehr als 700 Ziele
- Designtool für benutzerdefinierte Assays für auf GeneGlobe erhältliche bakterielle, Pilz- oder virale Ziele
- Farbstoffauswahl ermöglicht das Multiplexing mit bis zu 5 Zielen je Reaktion
- Einfacher und schneller dPCR-Workflow auf dem QIAcuity
- Kombination mikrobieller DNA- und viraler RNA-Ziele in einer Reaktion mit dem QIAcuity OneStep Advanced Probe Kit
Angaben zum Produkt
Mikrobielle Identifikation und mikrobielles Profiling sind von Interesse für viele Gebiete wie das Gesundheitswesen, Lebens- und Futtermitteltests sowie Umwelttests. Die mikrobielle Identifikation bestimmt die An- oder Abwesenheit eines Mikroorganismus in einer Probe, während das mikrobielle Profiling seine relative Expression unter zwei oder mehr experimentellen Bedingungen bestimmt, weshalb es eine Referenzprobe und einen Normalisator erfordert.
Unsere dPCR Microbial DNA Detection Assays zielen auf bakterielle, Pilz-, parasitische, virale, Antibiotikaresistenz- oder Virulenzfaktorgene ab. Für Bakterien zielt der Assay auf das 16S-rRNA-Gen und für Pilze auf ribosomale RNA-Gene ab. Das Portfolio besteht aus über 700 verschiedenen Assays, von denen 200 im Nasslabor für die dPCR getestet wurden. Für Ziele, die nicht von unserem Portfolio vorgefertigter mikrobieller dPCR-Assays abdeckt sind, bieten wir ein benutzerfreundliches Assay-Designtool für bakterielle, Pilz- und virale Ziele an.
Jeder Assay umfasst ein Primerpaar und eine Hydrolysesonde mit einem konfigurierbaren Fluorophor. Die verfügbaren Fluorophore sind FAM, HEX, ROX, TAMRA und Cy5. Diese können beliebig miteinander kombiniert werden und unterstützen so die Analyse von bis zu fünf verschiedenen Zielen in einer Multiplex-dPCR-Reaktion. Wir bieten auch im Labor getestete dPCR-5-plex-Assay-Pakete zur Analyse häufiger Ziele an; weitere Details finden Sie unter Anwendungen unten. Für die dPCR-Reaktion wird der Assay mit dem QIAcuity Probe PCR Kit (DNA-Ziele) oder dem QIAcuity OneStep Advanced Probe Kit (RNA-Ziele) kombiniert.
Der Assay wird zusammen mit dem QIAcuity Digital PCR System und den QIAcuity Nanoplates eingesetzt.
Möchten Sie Näheres über dieses Produkt erfahren und von einem unserer dPCR-Spezialisten kontaktiert werden? Registrieren Sie sich hier, und wir werden uns umgehend mit Ihnen in Verbindung setzen.
Leistung
Genauer und präziser Nachweis
Die dPCR Microbial DNA Detection Assays und das QIAcuity dPCR System ermöglichen eine genaue Quantifizierung auf Basis der digitalen PCR. Die Analyse von NIST-Referenzmaterial ergab die erwartete Konzentration von der Eingabemaske. Weitere Details siehe die Abbildung Quantifizierung des NIST-Referenzstandards 8376 unter Einsatz von gDNA von Shigella sonnei.
Leistung in Multiplex
Der Multiplex-dPCR-basierte Nachweis mikrobieller Ziele bietet eine flexible Einrichtung kleiner Panels in weniger Reaktionen, sodass mehr wertvolle Probe erhalten bleibt. Die Multiplexierung steigert auch den Analysedurchsatz, da sie weniger Nanoplatten-Wells erfordert und damit die Anzahl der Proben steigert, die pro Durchlauf analysiert werden können. Unsere dPCR Microbial DNA Detection Assays können für Multiplex-Analysen kombiniert werden.
Sie können Ihre eigenen Assays zur Multiplexierung kombinieren, indem Sie einzelne Assays mit verschiedenen Farbstoffen (FAM, HEX, TAMRA, ROX oder Cy5) bestellen. Oder wählen Sie aus unserem Angebot an 5-plex-Assay-Paketen, die bereits von unseren Wissenschaftlern im Nasslabor mittels dPCR getestet wurden. Andere Kombinationen der > 700 vorgefertigten Assays sollten in Ihrem eigenen Labor verifiziert werden.
Ein Vergleich zwischen Einzelplex- und Multiplex-Analysen unter Verwendung der dPCR Microbial DNA Detection Assays ergab eine ähnliche und hochpräzise Quantifizierung für alle Ziele (siehe Abbildung Mikrobieller Nachweis mittels Multiplex-dPCR auf dem QIAcuity). Zusätzlich zur präzisen Zielquantifizierung zeigt ein 5-Plex dPCR-Durchlauf auch einen hochspezifischen Nachweis von mikrobiellen Wasserpathogenen an (siehe Abbildung Nachweis von mikrobiellen Wasserpathogenen im 5-plex auf QIAcuity).
Prinzip
Die vorgefertigten Microbial DNA Detection Assays und die Custom dPCR Microbial Assays sind zur Verwendung in der digitalen PCR mit Nanoplatten vorgesehen. Jeder Assay basiert auf einer Endpunkt-PCR-Amplifikation einer speziesspezifischen genetischen Region des relevanten Mikroorganismus oder einer Region eines einzelnen mikrobiellen Gens. Das amplifizierte Produkt wird mithilfe zielspezifischer fluoreszierender Hydrolysesonden nachgewiesen, was die Spezifität des Assays verbessert.
Die dPCR Microbial DNA Detection Assays zum Nachweis bakterieller Spezies zielen auf die ribosomalen RNA-Gene – hauptsächlich das 16S-rRNA-Gen – ab und wurden unter Verwendung der GreenGenes-Datenbank für 16S-Sequenzen und von beim NCBI hinterlegten Typenstamm-DNA-Sequenzen entwickelt. Assays für Pilz-, Viren- und Metazoen-Spezies zielen auf unterschiedliche zielspezifische genetische Regionen ab, einschließlich ribosomaler RNA-Gene und anderer individueller Markergene, die alle beim NBCI hinterlegt sind. Verschiedene Datenbanken wurden für das Design der Assays für Antibiotikaresistenzgene (z. B. lahey.org, ARDB usw.) und Virulenzfaktorgene (z. B. VFDB) verwendet.
Die Custom dPCR Microbial Assays werden mit unserer Software zum Design benutzerdefinierter Assays erstellt. Diese basiert auf einem anspruchsvollen und gründlich getesteten Algorithmus, der spezifisch für mikrobielle Ziele entwickelt wurde. So wird gewährleistet, dass jedes Assay-Design strenge Leistungskriterien erfüllt und robuste, hochwertige Assays mit optimaler Sensitivität und Spezifität liefert.
Das Prinzip der dPCR-Reaktion in den Nanoplatten finden Sie hier beschrieben.
Verfahren
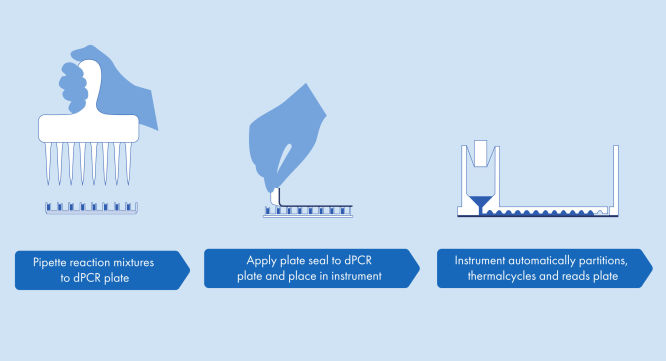
Das Protokoll der dPCR Microbial DNA Detection Assays und der Custom dPCR Microbial Assays ist einfach und kann in jedem Labor mit dem QIAcuity dPCR Gerät durchgeführt werden. DNA wird aus der Probe isoliert und dem gebrauchsfertigen QIAcuity Probe Mastermix und Microbial DNA-Free Water (UCP-Wasser) zugegeben. Dieses Gemisch wird in die einzelnen Wells der dPCR-Vorplatte aliquotiert, welche vordosierte Sets aus Primern und Hydrolysesonden enthalten. Die Reaktionsgemische werden aus der Vorplatte in die Wells einer dPCR Nanoplate überführt, welche dann versiegelt und in das QIAcuity dPCR Gerät überführt wird (siehe Abbildung Ein einfacher und schneller plattenbasierter Workflow). Die Partitionierungs-, Cycling- und Bildgebungsschritte werden unter Beachtung der voreingestellten Parameter vollständig durch das QIAcuity dPCR Gerät automatisiert. Abhängig vom Cycling-Protokoll können die Ergebnisse des dPCR-Laufs nach ~2 Stunden in der QIAcuity Software Suite analysiert werden (siehe Abbildung Mikrobielle dPCR in etwa 2 Stunden mit minimalem Zeitaufwand für den Benutzer).
Anwendungen
Die dPCR Microbial DNA Detection Assays sind bestens für den Nachweis von bakteriellen, Pilz- und viralen Spezies sowie mikrobiellen Antibiotikaresistenz- oder Virulenzfaktorgenen geeignet. Eine Vielzahl verschiedener Proben kann analysiert werden, z. B. Stuhl, Auswurf, Vaginalabstriche, Abwasser und mehr.
Zudem ermöglicht das Designtool für die Custom dPCR Microbial Assays das reibungslose Design von Assays für alle mikrobiellen Ziele von Interesse (Bakterien, Pilze, Viren), die nicht von unseren vorgefertigten dPCR Microbial DNA Detection Assays abgedeckt sind.
Laborgetestete dPCR-5-plex-Pakete
| Paket-ID | Anwendungsgebiet | Assay-Katalog nummern und Fluorophor* | Ziele (NCBI-Taxonomie-ID) |
| WW-001 | Abwasser 1 | DMA00278-F DMA00291-R DMA00340-H DMA00192-T DMA00344-C | Pseudomonas aeruginosa (287) Salmonella enterica (28901) Vibrio cholerae (666) Legionella pneumophila (446) Yersinia enterocolitica (630) |
| WW-002 | Abwasser 2 | DMA00340-H DMA00344-R DMA00199-C DMA00192-T DMA00710-F | Vibrio cholerae (666) Yersinia enterocolitica (630) Listeria monocytogenes (1639) Legionella pneumophila (446) Humanes Coronavirus SARS-CoV-2 (2697049) |
| WW-003 | Abwasser 3 | DMA00109-F DMA00192-H DMA00340-T DMA00194-R DMA00344-C | Clostridium perfringens (1502) Legionella pneumophila (446) Vibrio cholerae (666) Leptospira alexanderi (100053) Yersinia enterocolitica (630) |
| HM-001 | Humanes Mikrobiom 1 | DMA00148-F DMA00143-T DMA00024-R DMA00003-C DMA00150-H | Faecalibacterium prausnitzii (853) Eubacterium rectale (39491) Akkermansia muciniphila (239935) Acidaminococcus fermentans (951) Finegoldia magna (1260) |
| PB-001 | Probiotika 1 | DMA00177-F DMA00185-T DMA00061-C DMA00137-R DMA00320-H | Lactobacillus acidophilus (1579) Lactiplantibacillus plantarum (1590) Bifidobacterium bifidum (1681) Enterococcus faecium (1352) Streptococcus salivarius (1304) |
| RG-001 | Resistenzgene 1 | DMA00566-F DMA00542-H DMA00576-T DMA00574-R DMA00575-C | Fluorchinolon-Resistenzgen QnrS Klasse-D-Beta-Lactamase OXA-10-Gruppe Vancomycin-Resistenzgen vanB Tetracyclin-Effluxpumpengen tetA Tetracyclin-Effluxpumpengen tetB |
| RG-002 | Resistenzgene 2 | DMA00557-T DMA00528-R DMA00548-F DMA00587-H DMA00553-C | Fluorchinolon-Resistenzgen QepA Klasse-B-Beta-Lactamase blaVIM-1-Gruppe Klasse-D-Beta-Lactamase OXA-48-Gruppe Sulfonamid-Resistenzgen sul1 (43904) Klasse-D-Beta-Lactamase OXA-58-Gruppe |
| VG-001 | Virulenzgene 1 | DMA00614-F DMA00635-T DMA00677-H DMA00680-R DMA00597-C | Minor Fimbrial Subunit (fimH) Gamma-Hämolysin Komponente B (hlgB) Shiga-artiges Toxin 1 Untereinheit B, codiert innerhalb des Prophagen CP-933V Shiga-Toxin Untereinheit B; Rezeptorbindungsuntereinheit Akzessorisches Cholera-Enterotoxin (ace) |
| CP-001 | Cannabisproduktion 1 | DMA00278-F DMA00302-H DMA00365-R DMA00199-C | Pseudomonas aeruginosa (287) Staphylococcus aureus (1280) Aspergillus niger (5061) Listeria monocytogenes (1639) |
| HM-002 | Humanes Mikrobiom 2 | DMA00271-F DMA00017-T DMA00049-C DMA00142-H DMA00317-R | Prevotella oralis (28134) Actinomyces viscosus (1656) Bacteroides fragilis (817) Eubacterium infirmum (56774) Streptococcus oralis (1303) |
| RG-003 | Resistenzgene 3 | DMA00579-F DMA00577-T DMA00573-R DMA00502-H DMA00559-C | aac(6')-Ib vanC oprm QnrB-1-Gruppe CTX-M-1-Gruppe |
| VG-002 | Virulenzgene 2 | DMA00664-F DMA00677-H DMA00678-T DMA00642-R DMA00680-C | ply stx1B stx2A invA stxB |
| VG-003 | Virulenzgene 3 | DMA00688-F DMA00668-H DMA00596-T DMA00597-R DMA00611-C | wbkA ptxA ace (E. faecalis) ace (V. cholerae) efaA |
* F: FAM, H: HEX, T: TAMRA, R: ROX oder C: Cy5
Ergänzende Daten und Abbildungen
Ein einfacher und schneller plattenbasierter Workflow.
Partitionierung, Thermocycling und Bildgebung sind komplett in einem vollautomatischen Instrument integriert, das Ergebnisse in unter zwei Stunden liefert. Nanoplattenformate eignen sich zur automatisierten Probenvorbereitung, wodurch sich der Zeitaufwand weiter verringert.