miRCURY LNA miRNA Custom PCR Assay
Cat. No. / ID: 339317
Eigenschaften
- Designen Sie Ihre eigenen LNA-optimierten Primersets für die digitale PCR für jede miRNA
- Unübertroffene Sensitivität erlaubt miRNA-Quantifizierung anhand von nur 1 pg Gesamt-RNA
- Hohe Spezifität ermöglicht Unterscheidung zwischen eng verwandten miRNAs und zwischen reifer miRNA und Vorläufern
- Schnelles dPCR-Protokoll aus zwei Schritten dauert weniger als 3 Stunden
- Absolute Quantifizierung von miRNA-Expressionsänderungen mittels dPCR unter Verwendung des QIAcuity Gerätes und des QIAcuity EG PCR Kit
Angaben zum Produkt
miRCURY LNA miRNA Custom PCR Assays sind maßgeschneidert designte miRNA-PCR-Primersets, die eine höchst sensitive und spezifische miRNA-Quantifizierung erlauben. Sowohl Vorwärts- als auch Rückwärtsprimer für die PCR-Amplifikation sind miRNA-spezifisch und wurden mittels LNA-Technologie optimiert. Die Assays werden im Röhrchenformat bereitgestellt und enthalten ausreichend Primer für 166 Reaktionen in einer Nanoplate 8.5k (12 μl je Reaktion) oder 50 Reaktionen in einer Nanoplate 26k (40 μl je Reaktion). Der vorgelagerte cDNA-Syntheseschritt wird mit dem miRCURY LNA RT Kit durchgeführt, während die digitale PCR-Amplifikation mit einem QIAcuity EG PCR Kit auf dem QIAcuity Gerät erfolgt.
Sie benötigen ein Angebot für Ihr Forschungsprojekt oder möchten Ihr Projekt mit unserem Expertenteam besprechen? Kontaktieren Sie uns!
Leistung
Optimierte digitale PCR-Analyse mit den QIAcuity EG PCR Kits und dem QIAcuity Gerät
Die miRCURY LNA miRNA PCR Assays dienen der hochspezifischen Zielamplifikation. Die Quantifizierung kann mittels zweier PCR-Methoden erfolgen: qPCR mit Standard-Real-time-Thermocyclern und digitale PCR mit dem QIAcuity Gerät und dem QIAcuity EG PCR Kit. Nach der cDNA-Synthese mit dem miRCURY LNA RT Kit ermöglicht die digitale PCR eine hochpräzise Quantifizierung des Zielmoleküls, wobei selbst kleinste Expressionsänderungen bei geringsten Konzentrationen nachgewiesen werden. Ein Untergruppe von zentralen miRNAs wurde auf dem QIAcuity Gerät experimentell validiert.
LNA steigert die PCR-Leistung
Die miRCURY LNA miRNA PCR Assays wurden unter Einhaltung strenger Designkriterien und im Labor validierter Algorithmen entwickelt. Der strenge Assay-Auswahlprozess stellt sicher, dass jedes Primer-Set die zuverlässigsten und genauesten Ergebnisse bei höchster Spezifität und Effizienz liefert. Die Tm-Normierung und die LNA-Verstärkung verleihen den Primern eine höhere Bindungsaffinität als bei herkömmlichen DNA-Primern, was die Sensitivität und Spezifität des Assays deutlich erhöht.
Verglichen mit anderen miRNA-PCR-Systemen, die entweder Primer mit Haarnadelstruktur oder Standard-DNA-Primer verwenden, liefern die LNA-optimierten Primer eine signifikant höhere Sensitivität, besonders bei AT-reichen miRNAs.
Prinzip
Der Design-Prozess
Mit dem miRCURY LNA miRNA Custom PCR Assay Design Tool können Sie auf einfache Weise hochsensitive und hochspezifische LNA-optimierte PCR-Primersets für jede miRNA designen, für die kein vorgefertigtes Produkt verfügbar ist. Der fortgeschrittene Algorithmus wertet anhand von mehr als 60 verschiedenen Kriterien ca. 3.000 Primerpaar-Designs aus, um innerhalb weniger Minuten LNA-optimierte Primersets für Ihre miRNA zu finden. Das Tool wurde für miRNAs entwickelt, kann aber auch für andere kleine RNAs zwischen 14 und 27 Nukleotiden Länge verwendet werden.
Die Design-Kriterien umfassen:
- Optimierung der Schmelztemperaturen durch Variation der LNA-Verteilung der Primer, um eine hohe Amplifikationseffizienz und -spezifität zu gewährleisten.
- Berechnungen und Anpassungen der Scores für Selbsthybridisierung und Kreuzhybridisierung für effiziente PCR-Reaktionen.
- Anpassungen zur Vermeidung der Bildung potenzieller Primer–Dimere in der PCR-Reaktion.
- Intelligente Positionierung von LNA-Basen in den Primern basierend auf unserem umfassenden Wissen über das LNA-Oligonukleotid-Design.
- miRBase führt Suchen zur Identifizierung potenzieller miRNAs mit hoher Sequenzgleichheit durch. Dies gewährleistet, dass die Primersets hochspezifisch für die kleine RNA sind, für die sie entwickelt wurden.
Ein einzigartiges System für das miRNA-Profiling
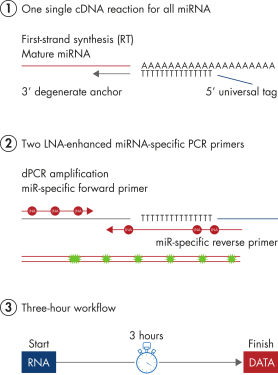
Die miRCURY LNA miRNA PCR Assays bieten durch Kombination aus universeller RT mit LNA-PCR-Amplifikation die beste auf dem microRNA-PCR-Markt verfügbare Kombination aus Leistung und Benutzerfreundlichkeit (siehe Abbildung Schematischer Überblick über das miRCURY LNA miRNA digitale PCR-System). Die universelle RT ermöglicht die Verwendung einer cDNA-Synthesereaktion des ersten Stranges als Template für mehrere miRNA-Real-time PCR-Assays. Dies spart wertvolle Probe sowie Zeit im Labor und reduziert technische Abweichungen. Darüber hinaus sind sowohl die Vorwärts- als auch die Rückwärtsprimer für die PCR-Amplifikation miRNA-spezifisch und wurden mittels LNA optimiert. Dadurch werden 1) eine außergewöhnliche Sensitivität und ein äußerst schwacher Hintergrund für eine genaue Quantifizierung sehr niedriger miRNA-Konzentrationen und 2) hochspezifische Assays, die die Unterscheidung zwischen eng verwandten miRNA-Sequenzen ermöglichen, erreicht.
Das Prinzip der dPCR-Reaktion in den QIAcuity Nanoplates finden Sie hier beschrieben.
Referenzgen-Assays zur Normalisierung
Für die dPCR sind fünf validierte Referenzgen-Primersets verfügbar. Sie ermöglichen eine qualitativ hochwertige Datennormalisierung und das Generieren zuverlässiger Daten für viele verschiedene Probentypen (siehe nachstehende Tabelle). Diese Kontrollprimersets zielen auf endogene, kleine, nicht codierende RNAs ab, die beständig und relativ stabil über verschiedene Gewebe hinweg exprimiert werden (siehe Abbildung Expressionsniveaus von Referenzgenen in verschiedenen menschlichen Geweben). Die Kontroll-Referenzgene bieten die Möglichkeit zur genauen und zuverlässigen Normalisierung über verschiedene miRNA-Expressionsniveaus hinweg. Weitere Informationen finden Sie auf der Seite miRCURY PCR Controls.
Liste der verfügbaren Referenzgen-Assays
| Produktbezeichnung | Zielorganismus | Alternativnamen | NCBI-Referenz |
| Control primer set, SNORD38B (hsa)* | hsa | U38B; RNU38B | NR_001457 |
| Control primer set, SNORD44 (hsa) | hsa | U44; RNU44 | NR_002750 |
| Control primer set, SNORD48 (hsa) | hsa | U48; RNU48 | NR_002745 |
| Control primer set, SNORD49A (hsa)* | hsa | U49; U49A; RNU49 | NR_002744 |
| Control primer set, SNORA66 (hsa) | hsa | U66; RNU66 | NR_002444 |
| Control primer set, 5SrRNA (hsa) | hsa, mmu | V00589 | |
| Control primer set, U6-snRNA (hsa, mmu)* | hsa, mmu, rno | U6 | X59362 |
| Control primer set, RNU5G-snRNA (mmu, hsa)* | mmu, hsa, rno | Rnu5a; U5a; Rnu5g | NR_002852 |
| Control primer set, RNU1A1 (mmu, hsa)* | mmu, hsa, rno | Rnu1a-1; Rnu1a1 | NR_004411 |
| Control primer set, SNORD65 (mmu) | mmu | U65; RNU65 | NR_028541 |
| Control primer set, SNORD68 (mmu) | mmu | U68; RNU68 | NR_028128 |
| Control primer set, SNORD110 (mmu) | mmu | U110; RNU110 | NR_028547 |
* Validiert zur Verwendung für die digitale PCR.
Abbildungen ansehen
Verfahren
Ein einfacher und schneller plattenbasierter Workflow.
Partitionierung, Thermocycling und Bildgebung sind komplett in einem vollautomatischen Instrument integriert, das Ergebnisse in unter zwei Stunden liefert. Die Nanoplattenformate eignen sich zur automatisierten Probenvorbereitung, wodurch sich der Zeitaufwand weiter verringert.
Genau wie bei qPCR-Experimenten umfasst die Probenvorbereitung die Überführung der verdünnten Proben und die Zugabe von Master-Mix und Primern in eine 96- oder 24-Well-Nanoplatte. Das System integriert Partitionierung, Thermocycling und Bildgebung in nur einem Vollautomaten, der den Benutzer in weniger als 3 Stunden von der Probe zum Ergebnis führt. Mit der Software Suite lassen sich Fernauswertungen durchführen, die die Konzentration der Zielsequenzen in Kopien pro Mikroliter liefern sowie Optionen für integrierte Qualitätskontrollen bieten.
Anwendungen
Die miRCURY LNA miRNA PCR Assays ermöglichen in Kombination mit dem QIAcuity Digital PCR System die digitale PCR-Analyse zur Quantifizierung reifer miRNA.
Ergänzende Daten und Abbildungen
Schematischer Überblick über das miRCURY LNA miRNA digitale PCR-System.