✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
QuantiTect Virus Kit (1000)
Cat. No. / ID: 211015
✓ Automatische Verarbeitung von Online-Bestellungen 24/7
✓ Sachkundiger und professioneller technischer und Produkt-Support
✓ Schnelle und zuverlässige (Nach-)Bestellung
Eigenschaften
- Hohe Sensitivität in Single- und Multiplex-Assays
- Nachweis von viraler RNA und/oder DNA in der gleichen Reaktion
- Eindeutiger Nachweis von schwach-positiven Signalen
- Schnelle, universelle 2-Schritt-Protokolle
- 5x Master-Mix für höhere Sensitivität bei größeren Probenmengen
Angaben zum Produkt
Leistung
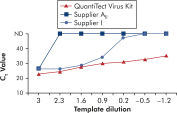
Die Amplifikation mit QuantiTect Virus Kits liefert steile sigmoidale Kurven über einen weiten Verdünnungsbereich, selbst bei geringen Template-Mengen mit hohen CT-Werten (siehe Abbildung „ Eindeutige Bestimmung der CT-Werte über einen breiten dynamischen Bereich“). Dies ermöglicht die genaue Bestimmung des CT-Werts bei der Quantifizierung viraler Nukleinsäuren mittels Real-time-PCR.
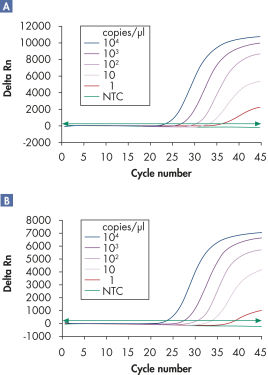
Multiplex-Assays ermöglichen den Nachweis mehrerer viraler RNA- und/oder DNA-Ziele sowie interner Kontrollen über einen breiten linearen Bereich ohne Verlust an Sensitivität (siehe Abbildungen „ Zuverlässiger Nachweis viraler RNA über einen breiten linearen Bereich“ und „ Verbesserter Nachweis geringer Mengen viraler RNA“).
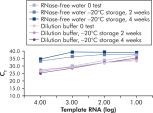
Der in den Kits enthaltene QuantiTect Nucleic Acid Dilution Buffer stabilisiert die RNA- und DNA-Standards während der Verdünnung und des Reaktionsansatzes und verhindert den Verlust von Nukleinsäuren durch Adsorption an Kunststoffoberflächen, wie z. B. Röhrchen oder Pipettenspitzen. Der Puffer ermöglicht eine zuverlässige Verdünnung von Standards, die zur Quantifizierung viraler Nukleinsäuren verwendet werden, und bietet so einen breiten linearen Bereich, der von niedrigen bis zu hohen CT-Werten reicht. Außerdem ermöglicht er eine längere Lagerung der Standards ohne Abbau (siehe Abbildung „ Zuverlässige Verdünnung und Lagerung von RNA-Standards“).
Abbildungen ansehen
Prinzip
Die QuantiTect Virus Kits ermöglichen auf Anhieb einen hoch sensitiven Nachweis viraler Nukleinsäuren in Singleplex- oder Multiplex-Assays (siehe Flussdiagramm „ QIAGEN Multiplex-Kits“). Der optimierte Master-Mix sorgt dafür, dass die PCR-Produkte in einer Multiplex-Reaktion mit der gleichen Effizienz und Sensitivität amplifiziert werden wie die PCR-Produkte in einer entsprechenden Singleplex-Amplifikationsreaktion.
Die Amplifikation von Kontroll- und Zielgenen in derselben Reaktion statt in getrennten Reaktionen erhöht die Zuverlässigkeit der Genquantifizierung, da Handhabungsfehler minimiert werden. Der QuantiTect Virus Buffer enthält eine ausgewogene Kombination aus K+- und NH4+-Ionen sowie den einzigartigen synthetischen Faktor MP. Zusammen sorgen sie für eine stabile und effiziente Hybridisierung der Primer und Sonden an das Nukleinsäure-Template und gewährleisten so eine hohe PCR-Effizienz (siehe Abbildung „ Einzigartiger PCR-Puffer“). Darüber hinaus sorgt die einzigartige Formulierung der Sensiscript Reverse Transcriptase für eine hoch sensitive reverse Transkription viraler RNA, während die HotStarTaq Plus DNA Polymerase einen stringenten Hot-Start gewährleistet und die Bildung unspezifischer Produkte verhindert.
| Kit-Komponente | Eigenschaft | Vorteile | |
|---|---|---|---|
| 5x QuantiTect Virus Master Mix | Konzentrierter Master-Mix | Hochkonzentriert und optimiert für den sensitiven Virusnachweis | Zur Erhöhung der Sensitivität können dem Assay größere Volumen an Template zugegeben werden |
| HotStarTaq Plus DNA Polymerase | Aktivierung innerhalb von 5 Minuten bei 95 °C | Ansatz der qPCR-Reaktionen bei Raumtemperatur | |
| QuantiTect Virus Buffer | Ausgewogene Kombination von NH4+- und K+-Ionen | Spezifische Primerhybridisierung für zuverlässige PCR-Ergebnisse | |
| Synthetischer Faktor MP | Zuverlässige Multiplex-Analyse von bis zu 4 Genen in einem Röhrchen | ||
| Zusätzliche Kit-Komponenten | QuantiTect Virus RT Mix | Enthält eine einzigartige Formulierung der Sensiscript Reverse Transcriptase | Optimiert für den hoch sensitiven Nachweis von viraler RNA |
| QuantiTect Nucleic Acid Dilution Buffer | Proprietäre Pufferformulierung für die Verdünnung und Lagerung von Nukleinsäurestandards. | Stabilisiert RNA- und DNA-Standards während der Verdünnung und des Reaktionsansatzes und verhindert den Verlust von Nukleinsäuren durch Adsorption an Kunststoffoberflächen, wie z. B. Röhrchen oder Pipettenspitzen |
Abbildungen ansehen
Verfahren
QuantiTect Virus Kits ermöglichen eine hoch sensitive Real-time-PCR-Analyse von viralen Nukleinsäuren (RNA und/oder DNA) und internen Kontrollen unter Verwendung sequenzspezifischer Sonden. Die Reaktionen können mit oder ohne reverse Transkription durchgeführt werden. Dies ermöglicht ein flexibles Design von Multiplex-Assays zum Nachweis von RNA-Zielen, DNA-Zielen oder sowohl RNA- als auch DNA-Zielen. Befolgen Sie das Protokoll im Handbuch für schnelle und zuverlässige Ergebnisse.
Die Kits sind mit oder ohne passiven Referenzfarbstoff ROX im Master-Mix erhältlich (siehe Tabelle).
| ROX-Farbstoff | Kit | Kompatible Thermocycler |
|---|---|---|
| Im Master-Mix enthalten | QuantiTect Virus Kit | Alle Thermocycler von Applied Biosystems außer Applied Biosystems 7500 |
| Wird in einem separaten Röhrchen geliefert | QuantiTect Virus +ROX Vial Kit | Applied Biosystems 7500 und Thermocycler von Bio-Rad, Cepheid, Eppendorf, QIAGEN, Roche, Agilent und anderen Herstellern |
Für schnelle und hoch sensitive Endpunkt-Ein-Schritt-RT-PCR-Anwendungen, einschließlich des Nachweises von Viren, empfehlen wir das QIAGEN OneStep RT-PCR Kit.
Anwendungen
Ergänzende Daten und Abbildungen
Eindeutige Bestimmung der CT-Werte über einen breiten dynamischen Bereich
Specifications
| Features | Specifications |
|---|---|
| Applications | Virusnachweis |
| SYBR Green I or sequence-specific probes | Sequenzspezifische Sonden |
| Real-time or endpoint | Real-time |
| Reaction type | Reverse Transkription und PCR |
| Thermal cycler | Die meisten Real-time-Thermocycler (außer Kapillar-Thermocycler, z. B. LightCycler® 1.x und 2.0) |
| Sample/target type | RNA- und/oder DNA-Ziele |
| With or without ROX | Erhältlich mit ROX im Master-Mix und ROX in einem separaten Röhrchen |
| Single or multiplex | Single- oder Multiplex |